
Reaktive Linker
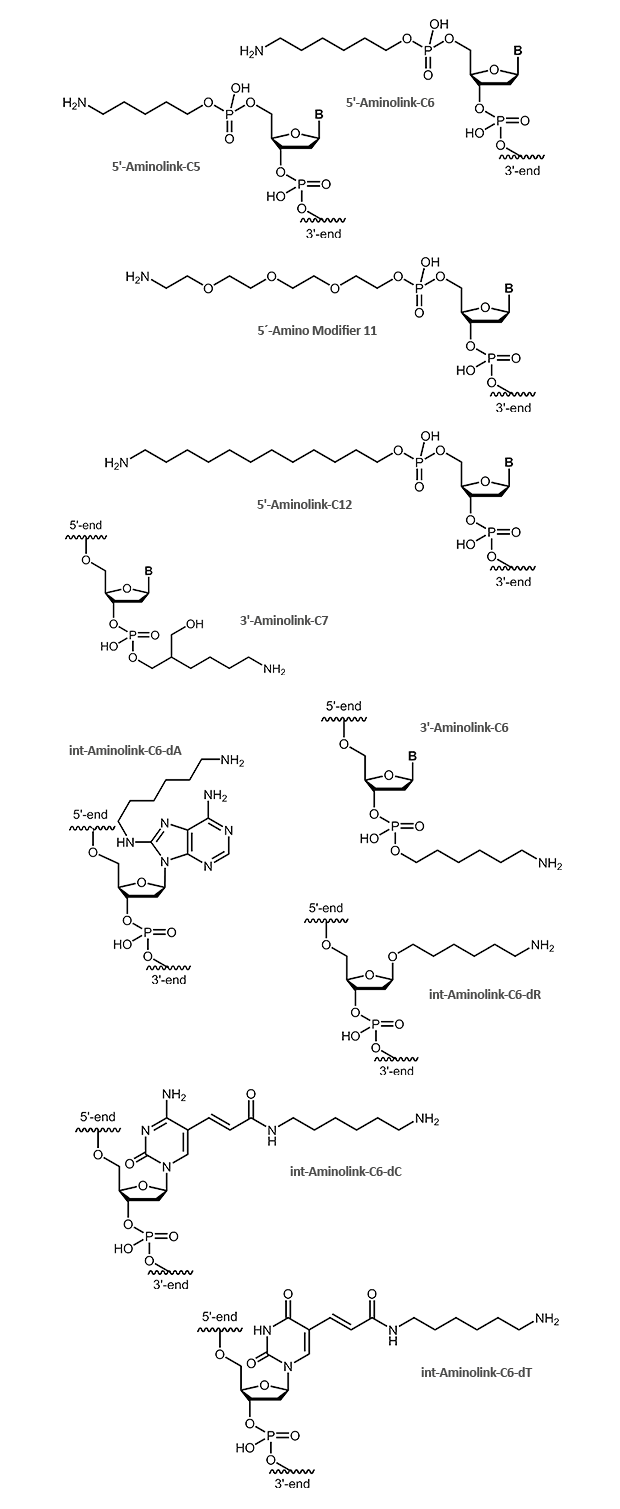
Aminolink
Aminolink
Ein Aminolink fügt eine endständige Aminogruppe (NH2) an einem Linker an. Die Aminogruppe bietet die Möglichkeit, über eine Amidbindung verschiedene weitere Moleküle zu koppeln, beispielsweise Farbstoffe oder auch Proteine wie HRP. Auch bei DNA-Arrays kommt der Aminolink zum Einsatz und wird hier genutzt, um die Oligonucleotide auf vorbereiteten Oberflächen anzuheften. Für verschiedene Anwendungen stehen unterschiedlich lange Linker zur Verfügung.
Ein Aminolink kann sowohl am 5´- als auch am 3´-Ende gekoppelt werden. Modifikationen am 3´-Terminus von Oligonucleotiden machen diese in der Regel resistenter gegen Exonuclease-Abbau. Darüber hinaus kann eine Aminogruppe auch direkt innerhalb des Oligonucleotids angeknüpft werden. Hierzu eignet sich besonders gut die Methyl-Gruppe am C5 von Thymidin-Nucleotiden. Statt dieser wird eine Aminogruppe über einen C6-Linker eingefügt. Dadurch wird gewährleistet, dass die Aminogruppe möglichst wenig mit der DNA wechselwirkt und ein so modifiziertes Oligonucleotid vergleichbar hybridisiert wie das entsprechende unmodifizierte.
Die Abbildungen zeigen das entschützte Endprodukt, wie es am fertigen Oligonucleotid zum Einsatz kommen kann.
 |
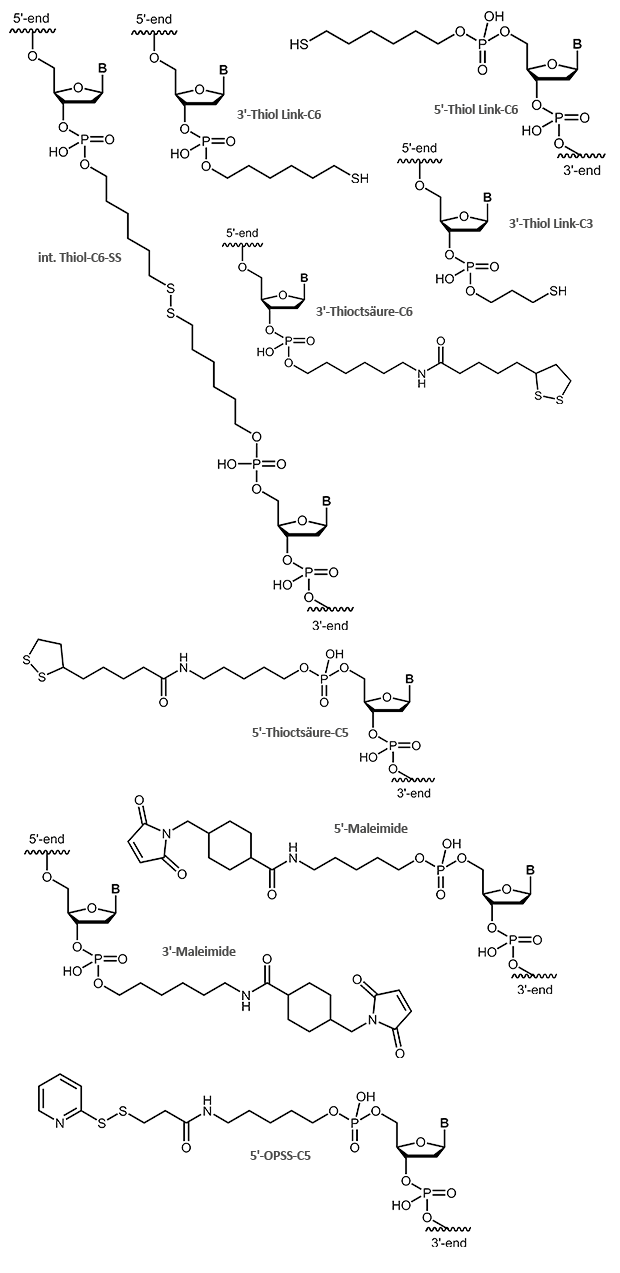
Thiol / Maleimid
Thiol und Maleimid
Endständige reaktive SH-Gruppen, z.B. zur Kopplung mit Maleimiden, können durch Thiolinker in Oligos eingeführt werden. Üblicherweise werden dabei C3- oder C6-Linker verwendet. Thiole und Maleimide reagieren leicht miteinander und ergeben eine stabile kovalente Verknüpfung beider Moleküle. Hierfür kann das Oligo entweder mit Thiol oder Maleimid aktiviert werden, je nachdem in welcher Form die Modifikation zur Verfügung steht. Beide Varianten sind möglich.
Ein interner Thiol-C6-SS-Linker erzeugt eine unter reduktiven Bedingungen spaltbare Stelle innerhalb einer Oligokette.
Zur Bindung von Oligonucleotiden an Goldoberflächen eignet sich neben einfachen Thiolen auch eine Thioctsäure-Modifikation; aufgrund der zwei Schwefelatome ist die Bindung zum Gold stärker.
OPSS, das Orthopyridyl Disulfid, ist eine endständige Modifikation, die mit einer SH-Gruppe (z.B. Cystein) eine stabile Disulfidbrücke ausbilden kann. Hierbei können an ein OPSS-modifiziertes Oligonucleotid Proteine, Peptide oder andere Biomoleküle, wie auch Oberflächen gekoppelt werden. Ebenso ist es möglich, die OPSS-Modifikation an die gewünschten Strukturen zu binden und das entsprechende Oligonucleotid mit einer Thiol-Verbindung zu modifizieren.
 |
Click-Chemie
Click-Chemie
Der Begriff „Click-Chemie“ beschreibt üblicherweise eine stark thermodynamisch begünstigte, schnelle Reaktion, die eine effiziente und selektive Verknüpfung zweier Moleküle ermöglicht. Im engeren Sinne versteht man unter „Click-Reaktion“ heutzutage eine Cycloadditionsreaktion zwischen einem Azid und einem Alkin. Es wird unterschieden zwischen der kupfer-katalysierten Azid-Alkin-Cycloaddition und der kupferfreien Click Chemie. Die Click-Reaktion verläuft sehr effizient in wässrigem Medium und ist daher für die Anknüpfung von Modifikationen an Biomoleküle oder die Verknüpfung von unterschiedlichen Biomolekülen geeignet. Sie ist weitgehend unempfindlich gegenüber anderen funktionellen Gruppen (z.B. Amino, Carboxy) und eröffnet daher viele Möglichkeiten.
Eine große Auswahl unserer Alkin- und Azidlinker, sowie der kupferfreien Varianten (DBCO, TCO, Tetrazin) finden Sie unter Click-Chemie.
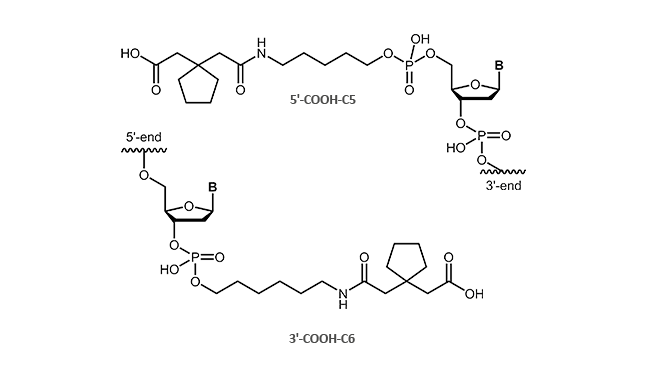
Carboxylinker
Carboxylinker
 |
Aldehyde
Aldehyde
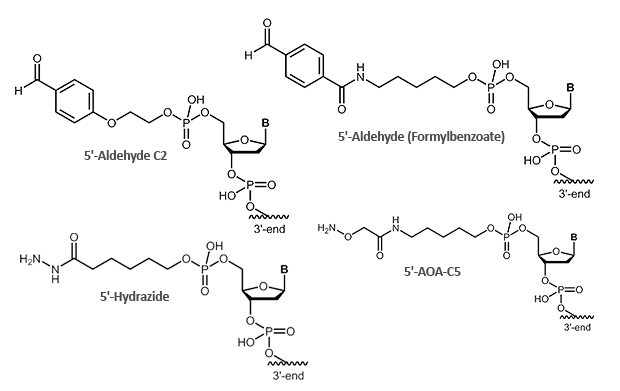
Aldehyde sind äußerst reaktive Verbindungen, die für die Verknüpfung von Oligonucleotiden mit anderen Biomolekülen genutzt werden können. Ganz allgemein reagieren hierbei Carbonylverbindungen (Aldehyde) mit Nukleophilen (Aminogruppen, Thiolverbindungen, etc.). Bei den Aminogruppen kann es sich um einfache Aminolinker (primäre und sekundäre Amine), aber auch um Hydrazin, Hydrazid oder Aminooxyverbindungen handeln, die mit Aldehyden reagieren können.
Aufgrund der teilweise komplex ablaufenden Reaktionen, werden Aldehyd-Reaktionen mit Aminooxyverbindungen (z.B. Aminooxyessigsäure) bevorzugt. Die Aminooxyessigsäure (AOA) reagiert mit Aldehyden zu stabilen Oximen.
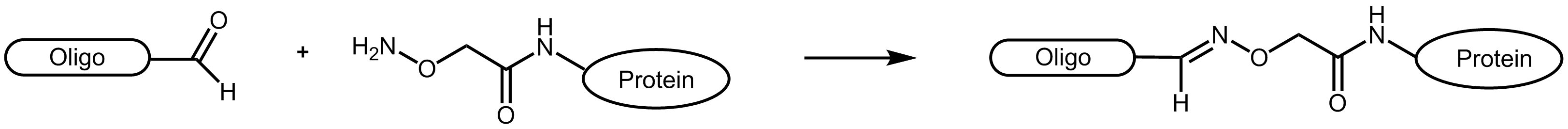
Abbildung 1: Reaktionsschemata eines Aldehyd-tragenden Oligonucleotids mit einem AOA-markierten Protein.
 |
Literatur:
1. Organic Chemistry, 4th edition. Carey FA; Chapter 22, p.858.
CBT / Cystein
Cyanobenzothiazol und Cystein – selektive Verknüpfung zweier Biomoleküle
Die Konjugationsreaktion zwischen Cyanobenzothiazol (CBT) und Aminothiolen, wie z.B. Cystein stellt einen weiteren effektiven Mechanismus dar, Biomoleküle kovalent miteinander zu verbinden. Das Konjugat dieser beiden Moleküle ergibt Luciferin, das Licht-generierende Substrat des Enzyms Luciferase, diese Reaktion ist seit langem als letzter Schritt der Luciferin-Synthese bekannt. 1
Die Reaktion von Aminothiolen und Cyanobenzothiazol läuft unter physiologischen Bedingungen spontan innerhalb lebender Zellen ab. 2
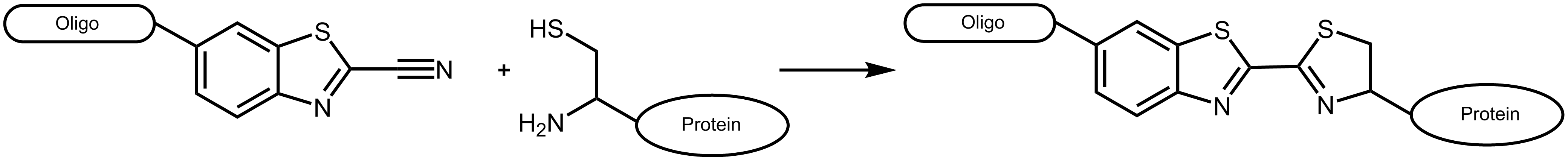
Abbildung 1: Reaktionsschema eines Cyanobenzothiazol-tragenden Oligonucleotids mit einem geeigneten Protein, das ein N-terminales Cystein trägt.
Cyanobenzothiazol reagiert dabei ausschließlich mit N-terminalen Cysteinen am Protein, nicht jedoch mit Cysteinen innerhalb der Aminosäuresequenz (Ausnahme siehe Ramil et al. 3). Da natürlich vorkommende Proteine für gewöhnlich kein terminales Cystein aufweisen, erlaubt dies ein effizientes und spezifisches Markieren entsprechend konstruierter Proteine.
Wir bieten Cyanobenzothiazol als 5´-Markierung am Oligonucleotid bis zu einer Länge von 40 Basen an.
Das Cyanobenzothiazol kann dabei entweder über NHS-Ester-Chemie an ein Oligo mit Aminolink [Struktur im Modifikationskatalog] oder über Click Chemie an ein Oligo mit einem Alkinlinker [Struktur im Modifikationskatalog] gekoppelt werden.
Neben der Kopplung an Proteine (Abbildung 3a), findet die CBT-Cystein-Reaktion auch im Bau von
Nanostrukturen 2 oder bei der Immobilisierung 4 Anwendung. Deshalb kann neben CBT-modifizierten Oligonucleotiden auch Cystein an das 5'-Ende von Oligos angehängt werden (Abbildung 3b, exemplarische Konjugation zweier Oligonucleotide).
a) b)

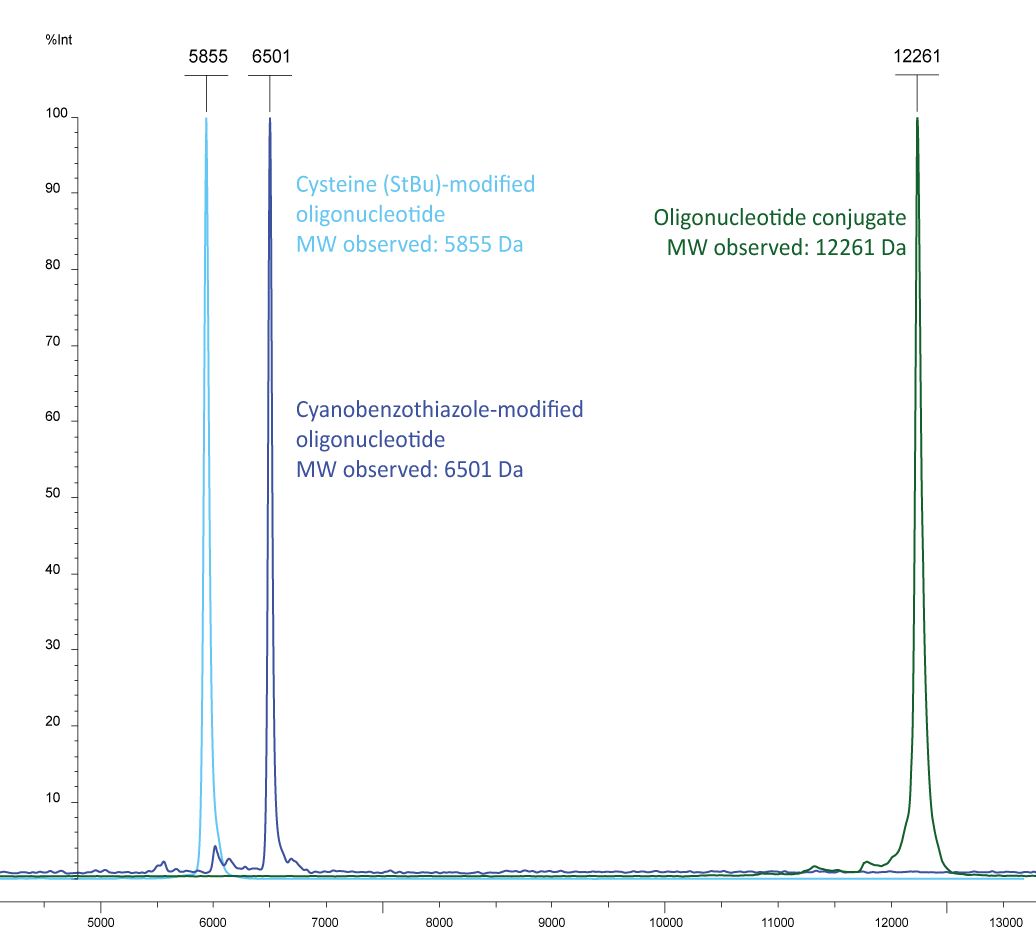
Abbildung 3a) zeigt die Maldi-Analyse einer Konjugationsreaktion zwischen einem Oligo und einem Peptid. Der blaue Peak stellt ein Cyanobenzothiazol-markiertes Oligo mit einer zusätzlichen Fluoreszenzmarkierung am 3´-Ende dar: EUB338-Sonde CBT-gctgcctcccgtaggagt-6-Fam (MW 6497). Dieses Oligo wurde mit einem Poly-Histidin (His6) mit N-terminalem Cystein konjugiert (MW 935). Der grüne Peak zeigt mit der Summe der beiden Molekulargewichte das Ergebnis der Konjugation.
Abbildung 3b): Die Reaktion der beiden Cystein- und CBT-markierten Oligos (in hellblau: Cystein-gctgcctcccgtaggagt, MW 5849, in dunkelblau: EUB338 CBT-gctgcctcccgtaggagt-6-Fam, MW 6497) ergibt das Konjugat mit einem summierten Molekulargewicht von 12261 (grüner Peak).
Zum Schutz der freien reaktiven Thiolgruppe liefern wir Cystein-markierte Oligos in geschützter Form (Cystein mit StBu Schutzgruppe). Vor weiteren Konjugationsschritten muss dem Kunjugationspuffer ein geeignetes Reduktionsmittel (z.B. TCEP) zugesetzt werden, um das Disulfid des Cysteins zu reduzieren und die Thiolgruppe freizusetzen.

Abbildung 4: Cystein mit einer StBu Schutzgruppe wird durch Behandlung mit TCEP zu freiem, reaktiven Cystein.
Literatur:
1. The structure and synthesis of firefly luciferin. White EH, McCapra F, Field GF, McElroy WD; J. Am. Chem. Soc. (1961), 83:2402–2403.
2. A biocompatible condensation reaction for controlled assembly of nanostructures in live cells. Liang G, Ren H, Rao J; Nat Chem. (2010), 2(1): 54–60.
3. Sequence-Specific 2-Cyanobenzothiazole Ligation. Ramil CP, An P, Yu Z, Lin Q; J Am Chem Soc. (2016), 15(6):829-35.
4. Site-specific immobilization of biomolecules by a biocompatible reaction between terminal cysteine and 2-cyanobenzothiazole. Wang P, Zhang CJ, Chen G, Na Z, Yao SQ, Sun H; Chem Commun (Camb). (2013); 49(77):8644-6.
Photoreaktive Linker
Photoreaktive Linker - Photocleavable Linker und Photocrosslinker
Die gezielte Freisetzung eines Moleküls sowie die Kopplung zweier Biomoleküle zu einem festgelegten Zeitpunkt ist für viele Anwendungen eine äußerst wünschenswerte Eigenschaft und findet im Bereich der Biokonjugation vermehrt Anwendung. Mithilfe eines extern-regulierbaren, nicht-invasiven Impulses (hier Licht) kann eine gezielte und kontrollierte Spaltung (Photocleavable Linker) bzw. Bindung (Photocrosslinker) hervorgerufen werden, sodass die beteiligten Biomoleküle zu einem definierten Zeitpunkt freigesetzt oder auch verknüpft werden können.
Unter photolabilen Linkerstrukturen versteht man im Allgemeinen eine Verbindung zwischen zwei Molekülen (Oligonucleotidsequenzen, Peptide, etc.) durch einen kurzen Linker, der nach Bestrahlung mit Licht einer bestimmten Wellenlänge (UV-Licht bei 300-400 nm) aktiviert und daraufhin gespalten werden kann.
Photoreaktive Linker sind äußerst selektiv und zeigen eine hohe Stabilität in verschiedenen chemischen Bedingungen. Die Energiezufuhr in Form von Licht stellt eine einfache, gut regulierbare „Schalterfunktion“ bei Konjugationen dar, da sie eine gezielte, zeitlich abstimmbare Reaktion (Spaltung oder Verknüpfung der Biomoleküle) ermöglicht.
Photocleavable Linker –lichtinduzierte Abspaltung markierter Oligonucleotide
Bei biomers.net erhalten Sie verschiedene UV-Licht-sensitive Linkerstrukturen:
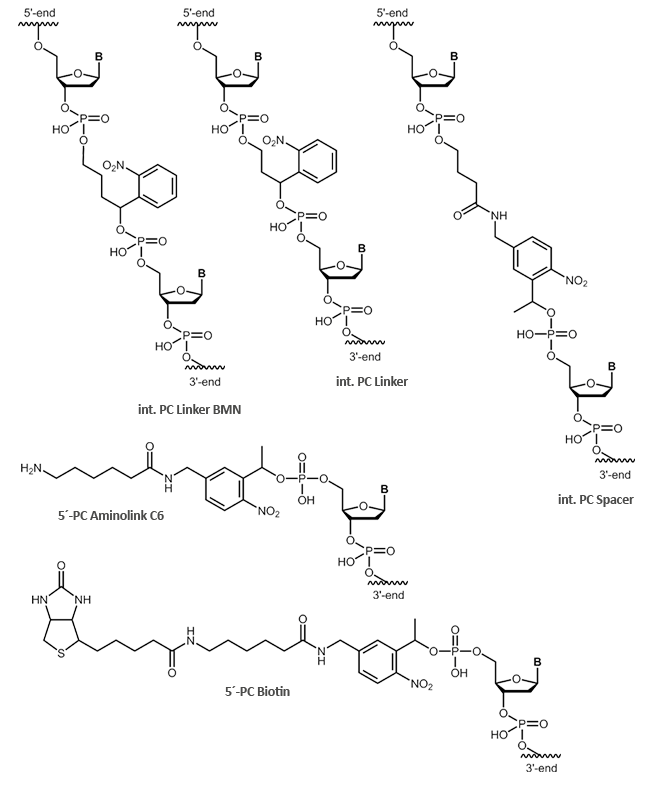
- PC Linker
- PC Spacer
- PC Biotin
- PC Aminolink C6
Neben den internen Modifikationen PC Linker und PC Spacer, sind auch abspaltbare, terminale Modifikationen, wie PC Biotin und der PC Aminolink C6, möglich. Die internen Modifikationen PC Linker und PC Spacer, können an jeder beliebigen Position innerhalb Ihrer Oligonucleotidsequenz einfügt werden.
Mithilfe von photocleavable Linkern (PC Linker) können organische Substrate an Festphasen oder andere Biomoleküle gekoppelt werden. Vor allem in sogenannten photo-induzierten Hybridisierungs-
anwendungen kommen PC Linker zum Einsatz.
Zur Lokalisierung und strukturellen Charakterisierung von Biomarkern werden sogenannte Tag-Mass Sonden verwendet. Der Tag-Mass basiert auf der indirekten Detektion von mRNA. Die spezifische Sonde bindet hierbei eine Targetsequenz und über einen gebundenen, abspaltbaren Tag mit bekannter Masse kann die Anwesenheit der mRNA indirekt nachgewiesen werden (spezifisches Maldi Imaging). Der Tag-Mass wird dabei über einen PC Linker an die Target-spezifische Sonde gebunden. Da die Wellenlänge zum Abspalten nahe der der eigentlichen Maldi-Analyse liegt, wird der Tag während der Messung im Maldi abgespalten und detektiert. Tag-Mass erlaubt somit eine schnelle und äußerst sensitive Messung des mRNA- und Protein-Expressionslevels und liefert hierdurch neue nützliche Informationen für Transkriptom-/ Proteom-Kolokalisationsstudien.
Zudem ermöglicht Maldi Imaging eine schnelle Detektion und Lokalisierung der Biomoleküle im Gewebe, sodass auf eine zeitaufwendige Extrahierung, Aufreinigung und Trennung vollständig verzichtet werden kann.
Die am häufigsten zum Einsatz kommenden PC Linker basieren auf einer Nitrobenzyl-Gruppe. Durch einen Lichtimpuls der Wellenlänge 300-400 nm wird die N=O Doppelbindung der Nitro-Gruppe aufgebrochen, woraus ein diradikaler angeregter Zustand entsteht, der während weiterer Reaktionsschritte schließlich in ein stabiles
2-Nitrosobenzaldehyd umgesetzt wird. Über die Dauer der Lichtexposition kann die Menge der abspaltbaren Biomoleküle reguliert werden.
Nach erfolgreicher Spaltung zwischen der Phosphatgruppe des Oligos und des PC Linkers erhält man jeweils ein
5´- und 3´-phosphoryliertes Oligonucleotid.
Photocleavable Linker erlauben einen hohen Grad an Chemoselektivität, da sie Forschern ermöglichen, räumliche, zeitliche und konzentrationsabhängige Variablen über die Zugabe von Licht zu steuern. Da die Abspaltung der Linkerstruktur keinerlei chemischer Zusätze bedarf, wird diese Methode häufig auch als „spurloser“ Prozess bezeichnet.
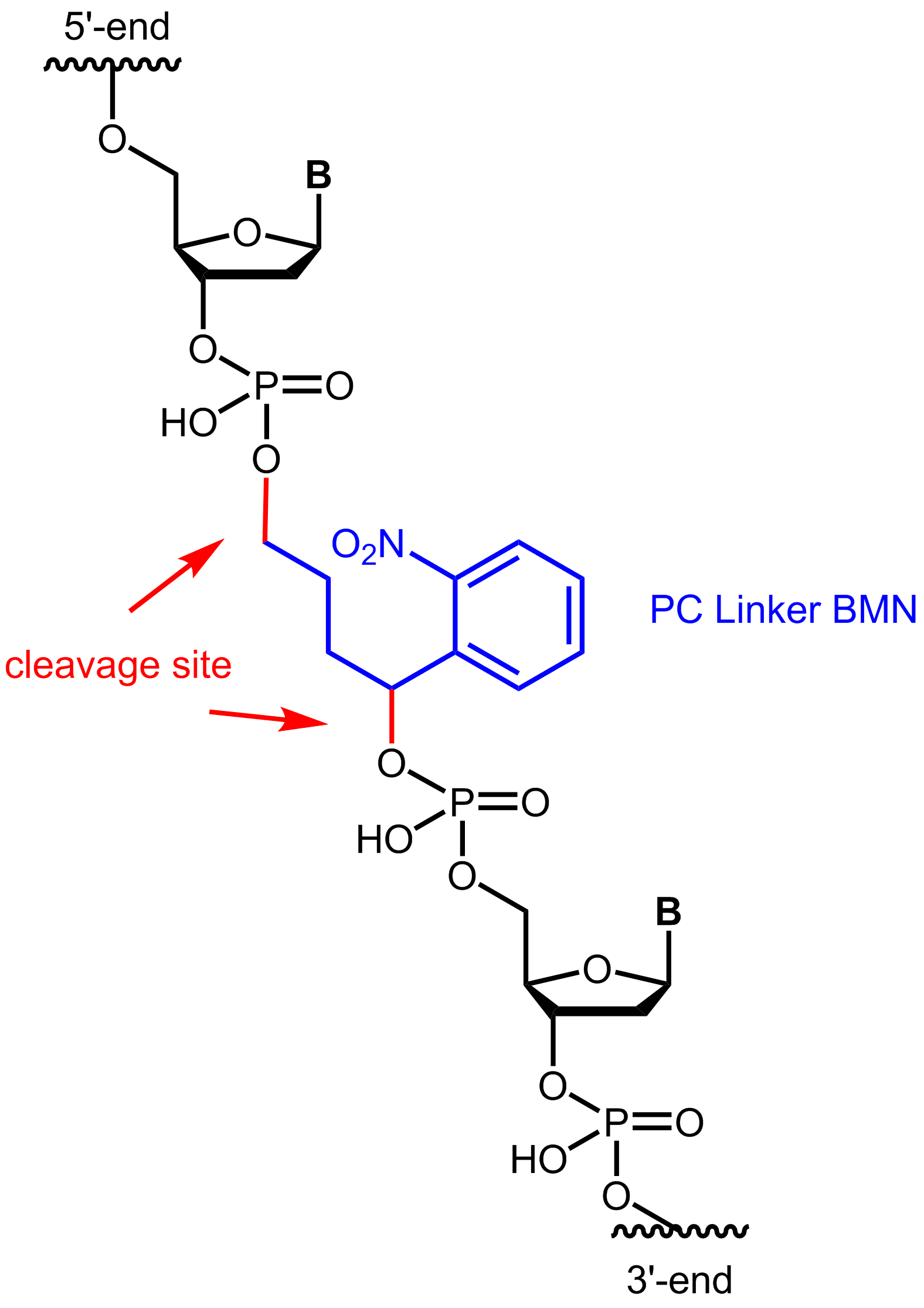
Abbildung 1: Struktur des internen photocleavable Linker BMN (PC Linker BMN) gebunden an ein Oligo.
Bereits die kurze Exposition mit energiereichem Licht durch eine Maldi-Messung reicht aus, um die Spaltprodukte eines Oligos mit internem PC Linker darzustellen.
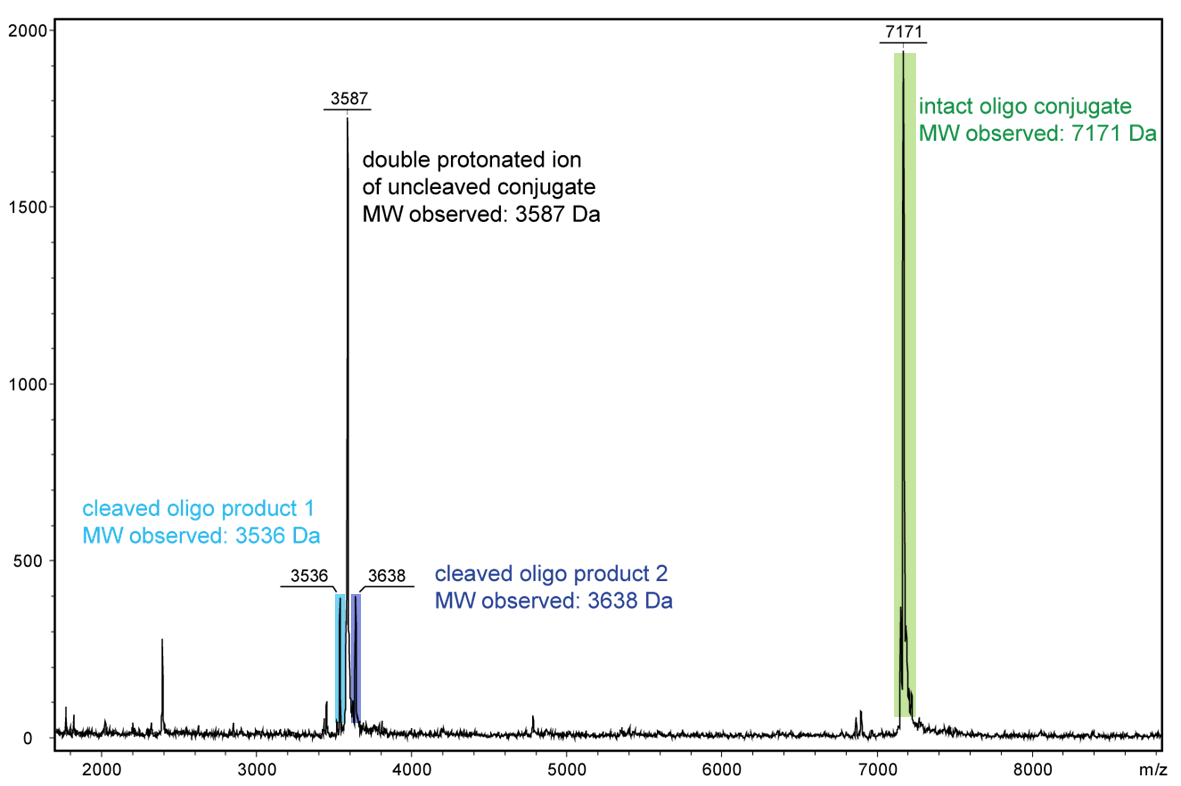
Abbildung 2 zeigt das Ergebnis der Maldi-Analyse eines dT20-Oligonucleotids mit einem internen photolabilen Linker (PC). Das vollständige Oligonucleotid mit einem Molekulargewicht von 7171 Da zeigt sich in dem großen Peak (grün unterlegt) auf der rechten Seite:
5’-Antraquinon-TTT TTT TTT T-PC-TTT TTT TTT T-BMN Q535-3’ (MW 7171 Da).
Die fast gleich großen Zerfallsprodukte durch den mittig in der Sequenz platzierten PC Linker sind blau unterlegt:
Produkt 1 5’-Phos-TTT TTT TTT T-BMN Q535-3’ (hellblau unterlegt, MW 3536 Da) und Produkt 2 5’-Antraquinon-TTT TTT TTT T-PC modified-3’ (dunkelblau unterlegt, MW 3638 Da).
Die hier dargestellten Spaltprodukte sind durch einen kurzen Laser-Impuls generiert worden, der überwiegende Anteil des Oligos ist ungespalten (grüner Peak). Der linke, große Peak zeigt das doppelt-protonierte Ion des nicht-gespaltenen Konjugats (MW 3587 Da).
a) 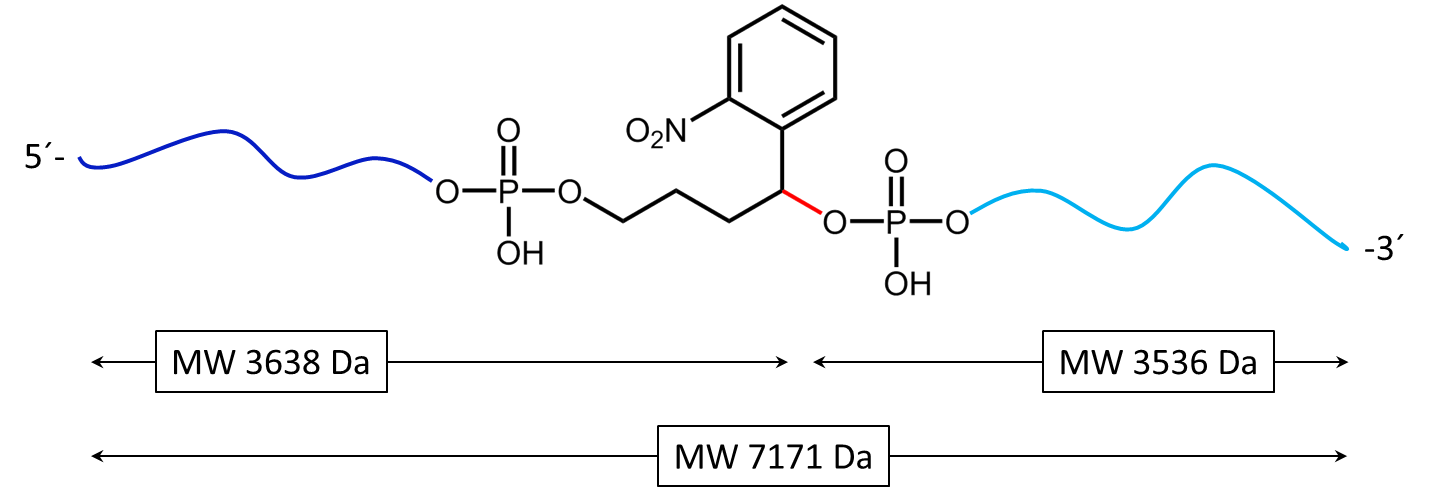
b) 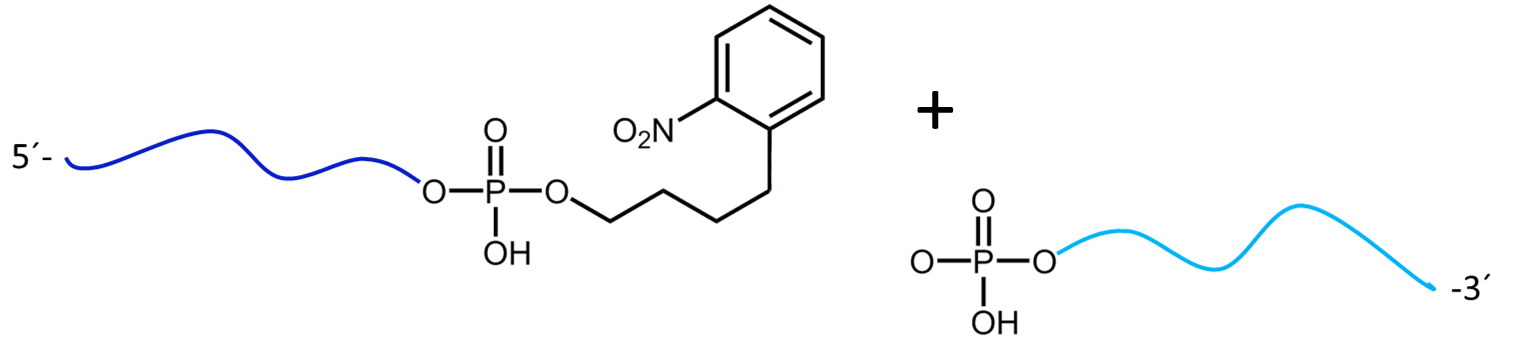
Abbildung 3a) zeigt die Spaltreaktion des Oligokonjugats bei Maldi-Laserbeschuss. Die Spaltstelle wird in rot dargestellt. In 3b) werden die entstehenden Spaltprodukte nach dem partiellen Zerfall des PC Linkers dargestellt.
Für eine vollständige Spaltung des photolabilen Linkers im Oligo wird länger exponiert, meist einige Minuten. Bei hinreichend langer Exposition zerfällt auch der Linker selbst. In der folgenden Abbildung 4 zeigt sich nach 30 minütiger Exposition ein weiteres Spaltprodukt, hierbei fällt der bereits gespaltene Linker vollständig von dem Oligo ab.
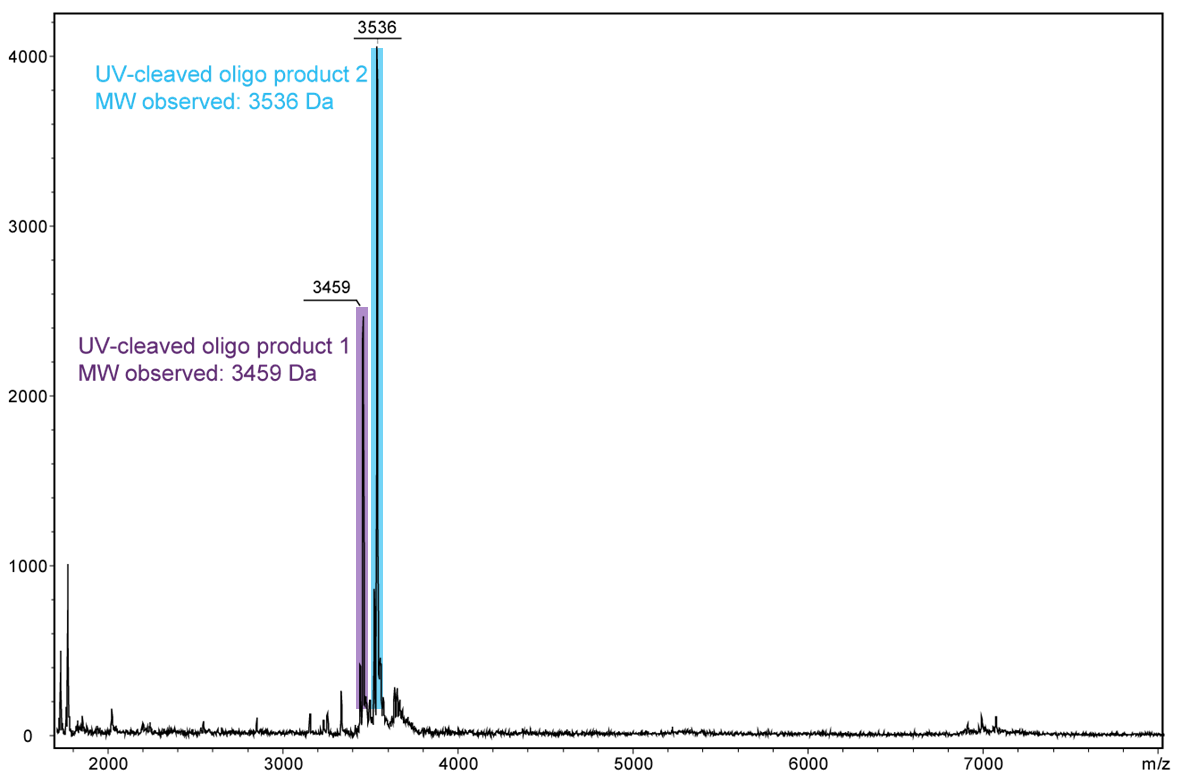
Abbildung 4) zeigt das Ergebnis der Maldi-Analyse des Oligokonjugats in wässriger Lösung nach Bestrahlung mit UV-Licht bei 365 nm für 30 min. Wird der PC Linker mit UV-Licht bestrahlt, zerfällt er und fällt vollständig vom Oligo ab. Das Spektrum zeigt die beiden Spaltprodukte des Konjugats: 5’-Antraquinon-TTT TTT TTT T-Phos-3’ (unterlegt in lila, 3459 Da) und 5’-Phos-TTT TTT TTT T-BMN Q535-3’ (unterlegt in hellblau, 3536 Da).
Das Oligokonjugat ist nach UV-Exposition vollständig gespalten.
a) 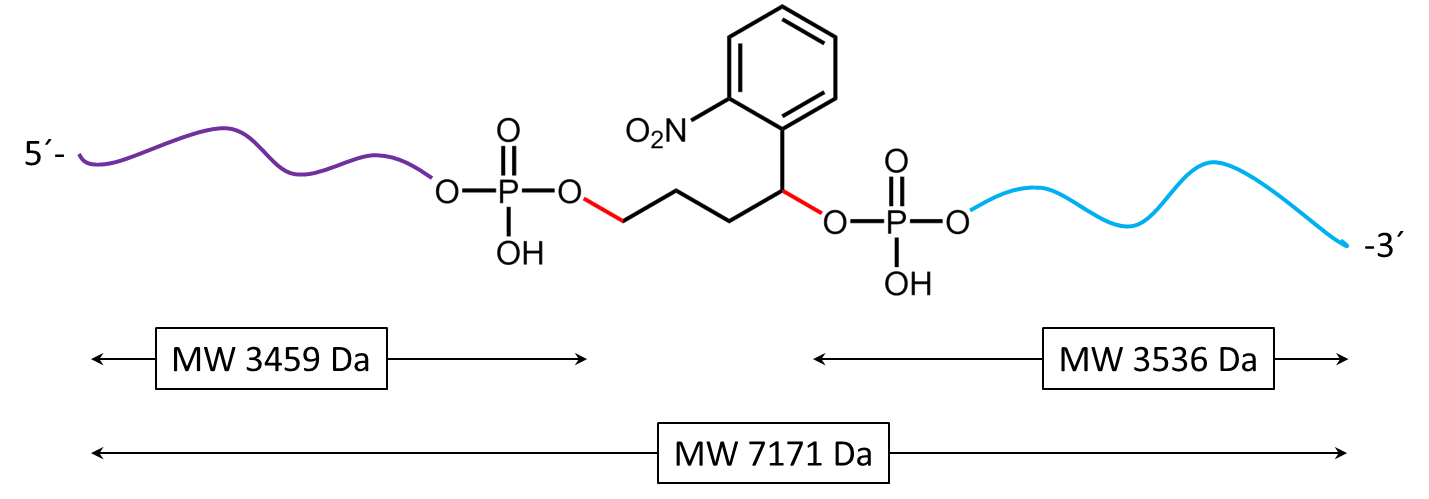
b)
Abbildung 5) zeigt die Spaltreaktion des Oligokonjugats bei UV-Bestrahlung. In rot werden die Spaltstellen dargestellt. 5b) stellt die entstehenden Spaltprodukte nach vollständigen Zerfall des PC Linkers dar.
Literatur:
1. Cleavable linkers in chemical biology. Leriche G, Chisholm L, Wagner A; Bioorg. Med. Chem. 20 (2012); 571–582.
2. Photocleavable linker for the patterning of bioactive molecules. Wegner SV, Sentürk OI, Spatz JP; Scientific Reports (2015), 5:18309.
3. Tag-Mass: Specific Molecular Imaging of Transcriptome and Proteome by Mass Spectrometry Based on Photocleavable Tag. Lemaire R, Stauber J, Wisztorski M, Van Camp C, Desmons A, Deschamps M, Proess G, Rudlof I, Woods AS, Day R, Salzet M, Fournier I; J Proteome Res. (2007), 6(6): 2057–2067.
4. Photocleavable peptide-DNA conjugates: synthesis and applications to DNA analysis using MALDI-MS. Olejnik J, Lüdemann HC, Krzymanska-Olejnik E, Berkenkamp S, Hillenkamp F, Rothschild KJ; Nucleic Acids Res. (1999), 27(23):4626-31.
5. Photocleavable biotin phosphoramidite for 5'-end-labeling, affinity purification and phosphorylation of synthetic oligonucleotides. Olejnik J, Krzymańska-Olejnik E, Rothschild KJ; Nucleic Acids Res. (1996), 24(2):361-6.
Photocrosslinker – Markierung und Crosslinking von Proteinen und Oligonucleotiden
Für ein effektives Crosslinking bieten wir Ihnen verschiedene photoreaktive Gruppen an, die am 5´-Ende des Oligonucleotids angebunden werden können:
- Benzophenon C5
- Diazirin C5
- Psoralen
- Azidobenzoat C5
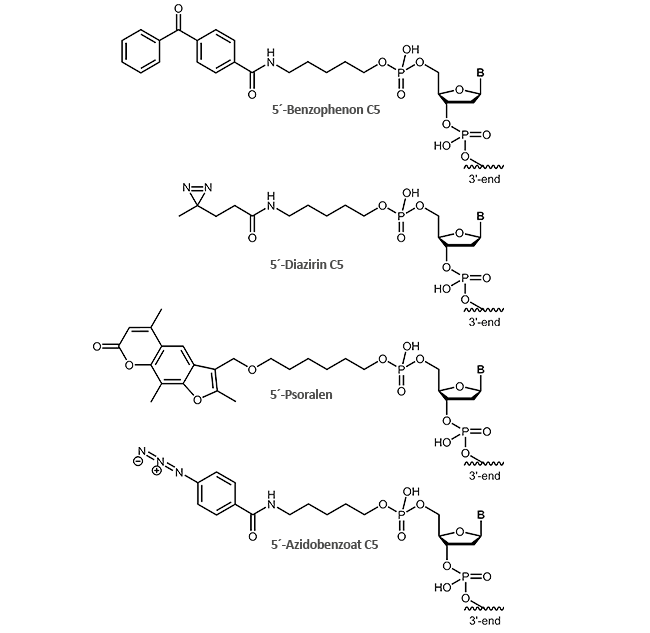
Wie der Name bereits vermuten lässt, werden Photocrosslinker durch Licht angeregt, um eine kovalente Verbindung zwischen zwei Molekülen herzustellen. Meist erfolgt die Lichtinduktion bei Wellenlängen nahe des UV-Bereichs (250-460 nm abhängig vom Linker) und kann zeitlich und räumlich genau gesteuert werden, z.B. nur in bestimmten Gewebe-Teilen oder zu einem definierten Entwicklungszeitpunkt.
Durch die Anbindung des Photocrosslinkers am Ende eines Oligonucleotids kann ein komplementärer DNA-Abschnitt „punktgenau“ angesteuert werden. Nach Photoinduktion entsteht eine kovalente Bindung zum gegenüberliegenden Strang. So kann beispielsweise eine Promotorregion und die damit verbundene Genexpression analysiert oder auch die endogenen Reparatur-Mechanismen erforscht werden, die Zellen entwickelt haben, um ihre DNA vor UV-Schäden zu schützen.
Als Alternative zu konventionellen Kopplungsstrategien bieten photoreaktive Linker ebenfalls die Möglichkeit funktionelle Gruppen an Oligonucleotide zu binden.
Weitverbreitet sind Photocrosslinker auch, um Proteine und DNA kovalent zu verknüpfen.
DNA-Protein-Kontakte sind wichtige Schaltstellen in Zellen, sodass eine genaue Kenntnis über die exakten Kontaktbereiche möglicherweise eine Modulation solcher Prozesse erlaubt.
Literatur:
1. Genetically encoded protein photocrosslinker with a transferable mass spectrometry-identifiable label. Yang Y, Song H, He D, Zhang S, Dai S, Lin S, Meng R, Wang C, Chen PR; Nat Commun. (2016), 7:12299. doi: 10.1038/ncomms12299.
2. Photolyase-like Repair of Psoralen-Crosslinked Nucleic Acids. Stafforst T, Hilvert D; Angew. Chem. Int. Ed. (2011), 50, 9483 –9486.
3. Sequence-specific photo-induced cross-linking of the two strands of double-helical DNA by a psoralen covalently linked to a triple helix-forming oligonucleotide. Takasugi M, Guendouz A, Chassignol M, Decout JL, Lhomme J, Thuong NT, Hélène C; Proc Natl Acad Sci U S A. (1991), 88(13):5602-6.
4. UV crosslinking of proteins to nucleic acids. Chodosh LA; Curr Protoc Mol Biol. (2001), Chapter 12:Unit 12.5. doi: 10.1002/0471142727.

