
Qualitätskontrolle
Maldi
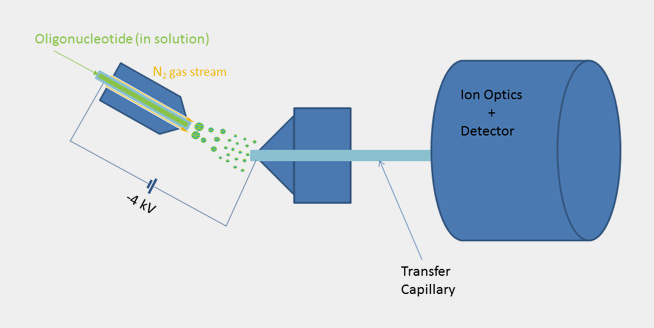
MaIdi-TOF Massenspektroskopie
biomers.net überprüft vor dem Versand die Integrität und Korrektheit jedes einzelnen Oligonucleotids (soweit technisch möglich). Mit Hilfe der Maldi-TOF Massenspektrometrie (matrix assisted laser desorption/ionisation; time of flight) lässt sich hinsichtlich seiner Molekülmasse (= Molekulargewicht) eine definitive Aussage über den Zustand eines Oligos treffen.
Das Prinzip der Methode beruht darauf, dass einzelne Moleküle in Abhängigkeit von ihrer Molekülmasse eine unterschiedlich lange Zeit benötigen, um eine bestimmte "Flugbahn" zurückzulegen (Time of Flight).
Durch die Verwendung eines entsprechenden Trägermaterials können Oligonucleotide mittels Laserbeschuss dazu angeregt werden, in einem ionisierten Zustand zu "fliegen" und im Vakuum eine definierte Strecke zurückzulegen. Die Zeit, die sie dazu benötigen, dient zur Berechnung des Molekulargewichts.
 |
Mittels Maldi lassen sich Unterschiede von einzelnen Wasserstoff-Atomen darstellen, im Bereich von Oligonucleotiden sind jedoch andere Größenordnungen interessant. Ein ganzes Nucleotid (im Zusammenhang von n-1-Produkten) beispielsweise hat ein Molekulargewicht von 300. Es kann also über das Maldi-Spektrogramm mit einem Blick erkannt werden, ob n-1-Produkte in nachweisbarer Menge vorhanden sind. Auch die erfolgreiche Kopplung von Modifikationen kann so überprüft werden; die meisten Farbstoffe zum Beispiel liegen in der Größenordnung von 500 - 800 Dalton.
Nur ein Oligo, das die vollständige Sequenz umfasst (und ggf. an dem die gewünschten Modifikationen erfolgreich gekoppelt sind) hat auch die errechnete Molekülmasse!
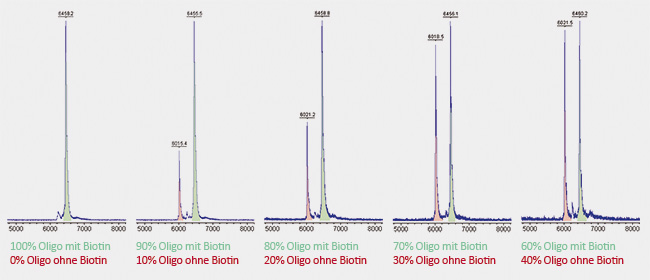
In der folgenden Abbildung ist dies dargestellt.
Hier wurden zwei identische Oligonucleotide hergestellt, jeweils 20 Basen lang, eines wurde 5'-terminal mit Biotin gekoppelt, das andere blieb ohne Biotin. Beide Oligonucleotide wurden auf gleiche Molarität gelöst und in verschiedenen Verhältnissen miteinander gemischt. Es zeigt sich bereits bei Zumischung von nur 10% unmodifiziertem Oligo ein signifikanter Peak (rot dargestellt).
Biotin hat auf Grund des verhältnismäßig langen Linkers ein Molekulargewicht von etwa 430 Da. Dem entsprechend ist der hier rot dargestellte Peak des unmodifizierten Oligo gegenüber dem grün dargestellten Peak des Biotin-tragenden Oligos um 430 Da nach links verschoben, das unmodifizierte Oligo ist "leichter" und kann die Flugstrecke zum Detektor in kürzerer Zeit überwinden.
 |
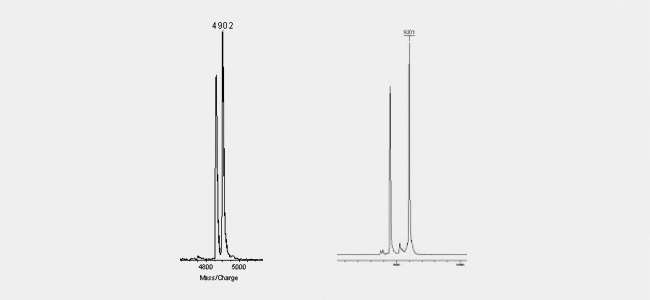
Wobble-Basen (Abbildung unten links) lassen sich ebenso erkennen wie die "Zerfallsprodukte", die bei der Messung von BHQ- oder BBQ-gekoppelten Oligos entstehen. Diese Quencher zerfallen durch den Laser-Beschuss in genau bekannte Teile, ein Teil verbleibt am Oligo, der andere fällt ab. Dieser Zerfall ist nicht vollständig, sodass im Allgemeinen zwei Peaks bei BHQ/BBQ-markierten Oligos zu sehen sind, die die korrekte Molekülmasse und die um das Abspalt-Fragment des Quenchers kleinere Molekülmasse haben (siehe Abbildung unten rechts).
 |
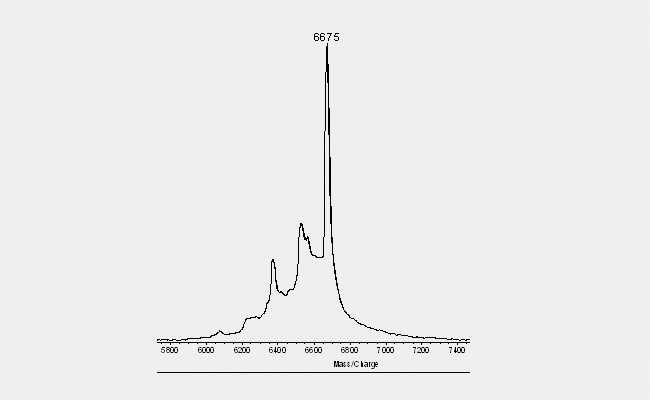
Eine weitere typische Erscheinung besonders bei purinreichen Oligos ist die so genannte De-Purinierung (siehe Abbildung unten). Dabei erscheint eine "Leiter" nach links kleiner werdender Peaks, die im Abstand von 150 Dalton dem Hauptpeak vorgelagert sind. Durch die Energie des Laserbeschusses werden in dem gemessen Aliquot einzelne Purine (MW: 150) aus dem Oligonucleotid "herausgeschossen", so dass eine gewisse Anzahl an Molekülen vorliegt, die um ein Purin "leichter" sind, eine kleinere Anzahl, denen zwei Purine fehlen und so weiter. Eine solche Leiter hat keinen Einfluss auf die Qualität des Oligonucleotids (der Beschuss betrifft nur das gemessene Aliquot).
 |
ESI
Electrospray Ionisation
Die ESI-MS ermöglicht die Massenbestimmung eines gelösten Oligonucleotids durch direktes Einbringen in die Ionenquelle oder alternativ über eine vorgeschaltete LC, die üblicherweise mit einer Reversed Phase Säule ausgestattet ist.
Wegen der milden Ionisation direkt aus der Lösung ist die ESI-MS gerade für längere Oligonucleotide sehr gut geeignet.
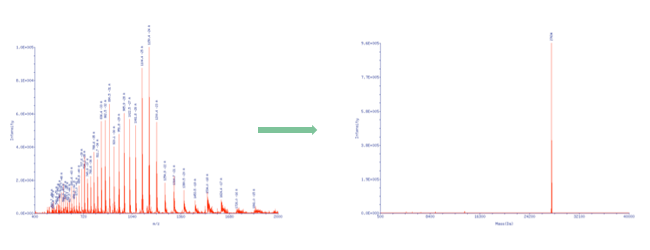
Im Rohspektrum findet man jedoch keinen diskreten Molekularpeak, sondern eine ganze Reihe von Signalen, im Positivmodus unterschiedlich geladene Kationen, im Negativmodus unterschiedlich geladene Anionen.
Im Falle von Oligonucleotiden bilden sich aus dem Sprühnebel der Probenlösung, die einen pH-Wert größer 7 aufweist, mehrfach geladene Anionen und damit ein Signalmuster, welches über eine mathematische Transformation den Rückschluss auf die eigentliche Molekülmasse ermöglicht.
|
 |
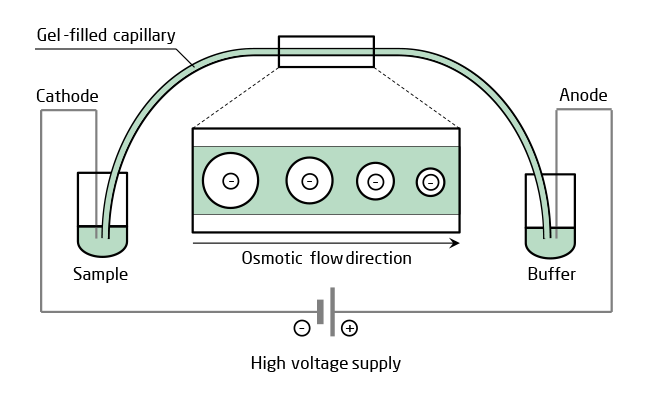
CGE
Capillary Gel Electrophoresis
Die CGE ist eine sehr empfindliche Analysenmethode, deren hohe Auflösung auf der Kombination von Elektrophorese und einem als Molekularsieb wirkenden Gel in einer miniaturisierten Kapillare beruht, welche problemlos temperiert werden kann.
In der Hochdurchsatz CE bietet sich die Verwendung einer Polymerlösung an Stelle eines vernetzten Gels an, da sich die Trennkapillare so zwischen den Läufen regenerieren lässt.
Wird nun eine kleine Menge des Analyten in eine mit Polymer gefüllte Kapillare injiziert und anschließend eine Hochspannung angelegt, so trennt sich die Oligonucleotidmischung im elektrischen Feld auf, da sich die Mobilität der Bestandteile wegen ihrer unterschiedlichen Größe und je nach Modifikation auch ihrer Ladung unterscheidet.
Über die UV-Detektion lässt sich die Anzahl und der Anteil der Nebenprodukte einschließlich des n-1 Oligonucleotids quantifizieren.
 |

