
Modifikationen für diverse Anwendungen
Gadolinium-DTPA
Gadolinium-DTPA (Gd-DTPA) modifizierte Oligonucleotide
Gadoliniumionen (Gd) verfügen über starke paramagnetische Eigenschaften und können dadurch die Spinrelaxation benachbarter Protonen beeinflussen. Diesen Effekt macht man sich in biochemischen NMR-Studien, jedoch vor allem in der Magnetresonanztomografie (MRT) im medizinischen Bereich zunutze, wo Gd-Komplexe als Kontrastmittel Anwendung finden.
Unter bestimmten Umständen können sich Oligonucleotid-Aptamere (DNA- oder RNA-Oligonucleotide) in spezifischen Zielgeweben anreichern. Sind diese Aptamere mit Gd-Ionen konjugiert, kann dies in entsprechenden bildgebenden Verfahren zu kontrastreicheren Aufnahmen führen.
Die Anbindung von Gadoliniumionen an Oligonucleotide erfolgt üblicherweise über die entsprechenden Chelatkomplexe (z.B. Gd-DTPA, Gd-DOTA). Praktischerweise wird dabei zunächst das Oligonucleotid mit einem Chelatbildner konjugiert. In einem nächsten Schritt werden dann die Metallionen (in diesem Fall Gd) „eingefangen“ und effizient gebunden, sodass ein äußerst stabiler Komplex entsteht.
Zur Markierung von Oligonucleotiden stehen verschiedene Chelatbildner und unterschiedliche Verknüpfungsstrategien zur Verfügung. Untenstehende Abbildung zeigt beispielhaft eine Gadolinium-DTPA Modifikation am 5´-Ende eines Oligonucleotids.
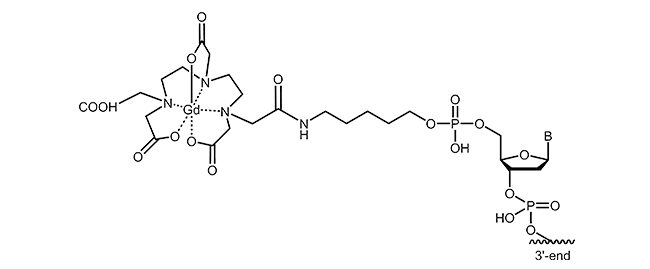
Literatur:
1. Gadolinium(III) Complexes with N-Alkyl-N-methylglucamine Surfactants Incorporated into Liposomes as Potential MRI Contrast Agents. Silva RS, Correia Duarte E, Ramos GS, Kock FVC, Diuk Andrade F, Frézard F, Colnago LA, Demicheli C; Bioinorganic Chemistry and Applications (2015), Volume 2015, Article ID 942147, 8 pages.
2. Characteristics of gadolinium-DTPA complex: a potential NMR contrast agent. Weinmann HJ, Brasch RC, Press WR, Wesbey GE. AJR Am J Roentgenol. (1984); 142(3):619-24.
3. Gadolinium-DTPA as a contrast agent in MRI: initial clinical experience in 20 patients. Carr DH, Brown J, Bydder GM, Steiner RE, Weinmann HJ, Speck U, Hall AS, Young IR; American Journal of Roentgenology. (1984); 143: 215-224. 10.2214/ajr.143.2.215.
4. Molecular MR Contrast Agents for the Detection of Cancer: Past and Present. Bogdanov A, Mazzanti ML; Semin Oncol. (2011); 38(1): 42–54.
5. Macromolecular MRI contrast agents: Structures, properties and applications. Tang J, Sheng Y, Hu H, Shen Y; Progress in Polymer Science (2013); Volume 38, Issues 3–4, Pages 462-502.
6. Gadolinium conjugated TiO2-DNA oligonucleotide nanoconjugates show prolonged intracellular retention period and T1-weighted contrast enhancement in Magnetic Resonance images. Paunesku T, Ke T, Dharmakumar R, Mascheri N, Wu A, Lai B, Vogt S, Maser J, Thurn K, Szolc-Kowalska B, Larson A, Bergan RC, Omary R, Li D, Lu Z-R, Woloschak GE; Nanomedicine (2008); 4(3): 201–207.
Puromycin
Puromycin
|
Puromycin ist ein Aminonucleosid-Antibiotikum, das aus dem Streptomycetenstamm |
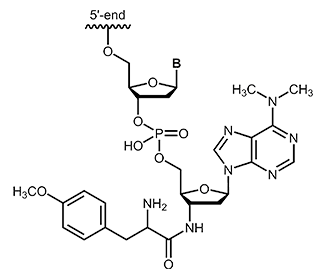 |
Puromycin erhalten Sie bei uns als 3´-Modifikation bis zu einer Oligolänge von max. 45 Basen.
Literatur:
1. Molecular cloning and characterization of gene encoding novel puromycin-inactivating enzyme from blasticidin S-producing Streptomyces morookaensis. Nishimura M, Ikeda K, Sugiyama M; J Biosci Bioeng. (2006), 101(1):63-9.
2. Identification and characterization of a drug-sensitive strain enables puromycin-based translational assays in Saccharomyces cerevisiae. Cary GA, Yoon SH, Torres CG, Wang K, Hays M, Ludlow C, Goodlett DR, Dudley AM; Yeast (2014), (5):167-78.
3. Puromycin oligonucleotides reveal steric restrictions for ribosome entry and multiple modes of translation inhibition. Starck SR, Roberts RW; RNA (2002), 8:890–903.
Acridinium Ester
Acridinium Ester
In der Molekularbiologie werden Acridinium Ester häufig bei der selektiven Markierung von Proteinen oder Nucleinsäuren verwendet1. Über eine kovalente Verknüpfung wird das Acridinium Ester an das entsprechende Target (DNA, Protein) gebunden2. Ihre hoch sensitive Chemilumineszenz, die sie in Anwesenheit von Wasserstoffperoxid freisetzen, ermöglicht ebenso den Einsatz in der klinischen Diagnostik. Im Gegensatz zu radioaktiv-markierter DNA stellen Acridinium Ester-Verknüpfungen eine sensitive, stabile und vor allem sichere Detektionsmethode für Immunoassays dar3.
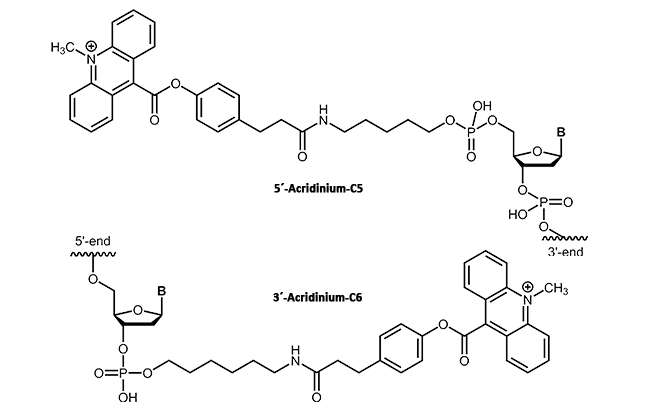 |
Bei biomers.net erhalten Sie Acridinium Ester als 3´- oder 5´-Modifikation bis zu einer Oligonucleotidlänge
von 45 Basen.
Literatur:
1. Acridinium ester chemiluminescence: pH dependent hydrolysis of reagents and flow injection analysis of hydrogen peroxide and glutamate. Stuever Kaltenbach M, Arnold MA; Microchimica Acta (1992), Volume 108, Issue 3-6, pp 205-219.
2. Acridinium ester-labelled DNA oligonucleotide probes. Septak M; J Biolumin Chemilumin. (1989), (1):351-6.
3. Acridinium esters as high-specific-activity labels in immunoassay. Weeks I, Beheshti I, McCapra F, Campbell AK, Woodhead JS; Clin Chem. (1983), (8):1474-9.
4-Carboxymethylanilin
Modifikationen für die Immobilisierung auf Oberflächen
4-Carboxymethylanilin (4-CMA) bietet die Möglichkeit, die daran gekoppelten Biomoleküle gezielt an entsprechend vorbereitete Oberflächen zu immobilisieren. Auf diese Weise ist die Bindung von Oligonucleotiden auch an leitende Oberflächen (z.B. Graphit) möglich, sodass Ströme gemessen werden und Strukturänderungen der gekoppelten DNA eine Art „Schalter-Wirkung“ haben können. Durch die Kopplung, wahlweise an den 5´- oder 3´-Terminus von Oligonucleotiden, werden die Moleküle in der gewünschten Orientierung kovalent an die Oberfläche gebunden.
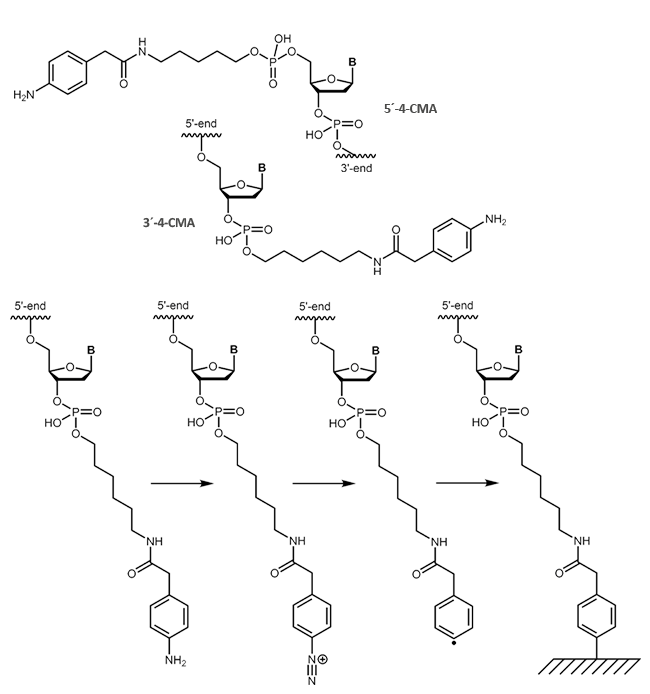 |
Literatur:
- Diazonium-Protein Adducts for Graphite Electrode Microarrays Modification: Direct and Addressed Electrochemical Immobilization. Corgier BP, Marquette CA, Blum LJ; Journal of the American Chemical Society, (2005), 127, 18328-18332.
- A versatile method for direct and covalent immobilisation of DNA and proteins on biochips. Corgier BP, Laurent A, Perriat P, Blum LJ, Marquette CA; Angewandte Chemie International (2007), 46, 4108-4110.
- On-Chip Chemiluminescent Signal Enhancement using Nanostructured Gold-Modified Carbon Microarrays. Corgier BP, Li F, Blum LJ, Marquette CA; Langmuir (2007), 23(16), 8619-8623.
Methacrylamid
Immobilisierung an Oberflächen über Methacrylamid-modifizierte Oligonucleotide
Methacrylamid-modifizierte oder auch bekannt als Acrydite-modifizierte Oligonucleotide sind seit einigen Jahren ein fester Bestandteil in der molekularbiologischen Forschung. Über die Polymerisierung freier Acrylsäure-Monomere (Bildung Polyacrylamiden) oder die Kopplung mit Thiol können Methacrylamid-modifizierte Oligonucleotide kovalent an Oberflächen gebunden werden. Über diese schnelle und einfache Anheftung an Oberflächen können gewünschte DNA-Einzelstränge immobilisiert, angereichert, identifiziert oder auch aufgereinigt werden.
Ein bekanntes Beispiel hierfür sind Biochips (Microarrays), bei denen die Methacrylamid-modifizierten Oligonucleotide auf eine Thiol-markierte Glasoberfläche gebunden werden und somit für einzelsträngige DNA zugänglich sind.
Auf diese Weise ergibt sich eine Vielzahl von möglichen Anwendungen:
- Überwachung der mRNA Expression
- Sequenzierung von DNA
- Genotypisierung
- Identifizierung von SNP
- Detektion von Viren, Bakterien und anderen Pathogenen
biomers.net bietet 5´-und 3´- Methacrylamid-modifizierte DNA-Oligonucleotide an.
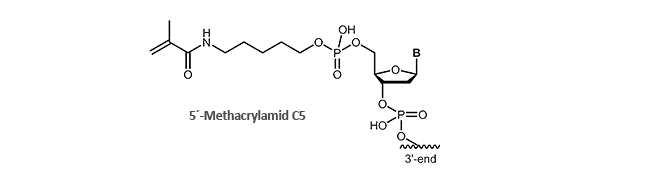
Literatur:
1. Immobilization of acrylamide-modified oligonucleotides by co-polymerization. Rehman FN, Audeh M, Abrams ES, Hammond PW, Kenney M, Boles TC; N. Acids Research. 27, (1998), Bd. 2, 649-655.
2. Mutation typing using electrophoresis and gel-immobilized Acrydite probes. Kenney M, Ray S, Boles TC; Biotechniques (1998), (3):516-21.
Gemcitabin
Oligonucleotide mit dem Zytostatikum Gemcitabin - dFdC
Gemcitabin (2´,2´-Di-Fluoro-Desoxycytidin, dFdC) ist ein Analogon des Pyrimidin-Nucleosids Desoxycytidin (dC). Gemcitabin unterscheidet sich von diesem durch zwei Fluor-Atome (anstelle zweier Wasserstoffatome) an der 2´-Position des Zuckers.
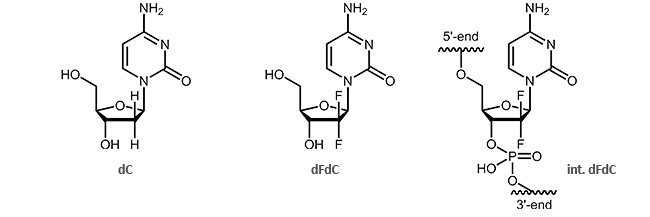
Diese zwei zusätzlichen Fluor-Atome führen zu einer Blockierung der DNA-Replikation.1,2 Interessant ist hierbei besonders, dass nach dem Einbau von Gemcitabin die DNA noch um ein weiteres Nucleotid verlängert wird. Danach bricht die Synthese ab und wird blockiert, was den Tod der Zelle zur Folge hat. Dieser Ablauf wird als „masked chain termination“ bezeichnet, da das letzte Nucleotid nach einem dFdC dieses vor Detektion und Abbau durch Exonucleasen oder DNA-Reparaturenzyme maskiert.3
Aufgrund seiner Wirkung als Zytostatikum wird Gemcitabin in der Chemotherapie als Anti-Tumor-Präparat eingesetzt.3
biomers.net bietet jetzt dFdC für den Einbau in Oligonucloetiden an!
Die interne Kopplung ist an jeder beliebigen Position innerhalb eines Oligonucleotids möglich, auch mehrfache Kopplungen sind herstellbar. Kombinationen mit anderen Modifikationen auf Anfrage.
Literatur:
1. 2',2'-Difluoro-deoxycytidine (gemcitabine) incorporation into RNA and DNA of tumour cell lines. Ruiz van Haperen VW, Veerman G, Vermorken JB, Peters GJ; Biochem Pharmacol. (1993); 46(4):762-6.
2. Quantification of gemcitabine incorporation into human DNA by LC/MS/MS as a surrogate measure for target engagement. Wickremsinhe ER, Lutzke BS, Jones BR, Schultz GA, Freeman AB, Pratt SE, Bones AM, Ackermann BL; Anal Chem. (2010); 82(15):6576-83. doi: 10.1021/ac100984h.
3. DNA Repair in Cancer Therapy: Molecular Targets and Clinical Applications. Kelley MR; Academic Press (2012), 95-98.
4. Synthesis and restriction enzyme analysis of oligodeoxyribonucleotides containing the anti-cancer drug 2',2'-difluoro-2'-deoxycytidine. Richardson FC, Richardson KK, Kroin JS, Hertel LW; Nucleic Acids Res. (1992); 20(7): 1763–1768.
CoA
Oligonucleotid-Coenzym A-Konjugate
Viele Forschungsgebiete erfordern eine gezielte Verknüpfung von möglichst nativen Proteinen mit anderen Bausteinen, die beispielsweise deren Transportverhalten beeinflussen oder eine spezifische Immobilisierung ermöglichen. Durch die Verwendung kleiner 'Tags', die idealerweise an die zu untersuchenden Proteine fusioniert werden können, ohne jedoch dabei deren Faltung oder Funktion zu beeinträchtigen, können Proteine nahezu beliebig kombiniert werden. Ein hochflexibles System macht sich die selektive Verknüpfung des sogenannten ybbR-Tags mit Coenzym A zunutze.
Wir freuen uns, Ihnen nun Coenzym A an Oligos gekoppelt anbieten zu können. Damit ergeben sich vielfältige Möglichkeiten von DNA-Protein-Chimären.
Ein eindrucksvolles Beispiel finden Sie unter:
Protein–DNA Chimeras for Nano Assembly (Pippig et al., ACS Nano, 2014)
Fragen Sie... gern diskutieren wir Ihr individuelles Projekt!
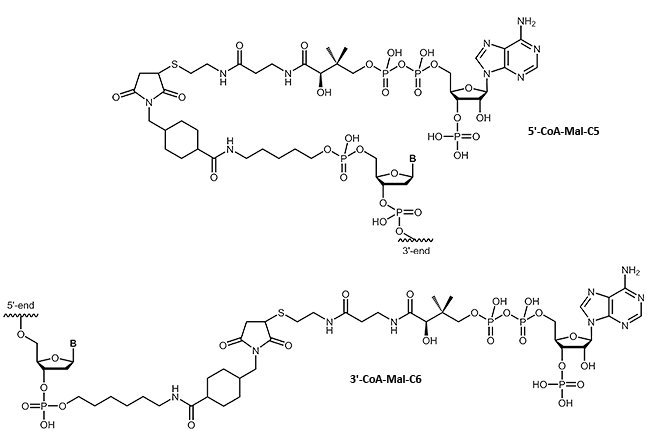 |
Literatur:
- Protein-DNA Chimeras for Nano Assembly. Pippig DA, Baumann F, Strackharn M, Aschenbrenner D, Gaub HE; ACS Nano (2014), 8 (7), pp 6551–6555.
- The Ribosome Modulates Nascent Protein Folding. Kaiser CM, Goldman DH, Chodera JD, Tinoco Jr. I, Bustamante C; Science (2011), 334(6063): 1723–1727. doi:10.1126/science.1209740.

