
DNA-Oligonucleotide
Standardscales für Oligonucleotide
Standard-Oligonucleotide bis 140 Basen
Standard-Oligonucleotide erhalten Sie von biomers.net in vier unterschiedlichen Scales. Hier eine Kurzübersicht zu den angebotenen Längen, Mengen und Reinigungen unserer Oligonucleotide:
| Synthese-Scale1 | XS 0,02 µmol |
S 0,05 µmol |
M 0,2 µmol |
L 1,0 µmol |
|---|---|---|---|---|
| Länge (Basen) | 12-60 | 12-140 | 6-140 | 6-140 |
| Wobbles | - | ja | ja | ja |
| Reinigungen2 | ||||
| Kartusche | ja | ja | - | - |
| HPLC | - | ja | ja | ja |
| PAGE | - | ja | ja | ja |
| Ausbeuten3 | ||||
| Kartusche/HPLC | > 2 OD | > 3 OD | > 10 OD | > 30 OD |
| PAGE | - | > 1 OD | > 3 OD | > 10 OD |
| Qualitätskontrolle4 | ||||
| Maldi | ja | ja | ja | ja |
1. Der Synthese-Scale bezeichnet die Menge an Startmaterial und nicht die Ausbeute an fertigem Oligo. Aufgrund der vielen Synthese-, Aufarbeitungs- und Reinigungsschritte ist die Endausbeute deutlich geringer als die Startmenge der Oligonucleotide.
2. Sämtliche Oligos werden mit mindestens Kartuschenreinigung ausgeliefert. Ungereinigte oder nur entsalzte Oligos bieten wir nicht an. Für Oligonucleotide mit einer Länge von über 60 Basen ist in den Scales S-L eine HPLC- oder PAGE-Reinigung zwingend erforderlich.
3. Die angegebene Ausbeute ist die garantierte Liefermenge für ein gemischtes 20mer DNA-Oligonucleotid. Für deutlich kürzere bzw. längere Oligos und Homopolymere kann die Liefermenge abweichen. Erfahrungsgemäß werden in vielen Fällen deutlich höhere Ausbeuten erzielt. Selbstverständlich senden wir Ihnen dann die gesamte Menge zu.
4. Jedes Oligonucleotid bis zu einer Länge von 50 Basen wird routinemäßig mit Maldi-Massenspektrometrie qualitätskontrolliert.
Kurze bis extra lange Oligos
2 - 5mer Oligonucleotide
Der eigentliche Syntheseprozess ist für nahezu alle Oligonucleotidlängen identisch. Aufgrund der geringen Molekülgröße unterscheiden sich sehr kurze Oligos allerdings bei der Isolierung und Reinigung deutlich von längeren Oligos. Wir bieten für Di-, Tri-, Tetra- und Pentamer-Oligonucleotide spezielle Synthese-Scales an, die die entsprechend angepasste Reinigung beinhalten:
| Synthese-Scale | DL2 | DL3 | DL4 | DL5 |
|---|---|---|---|---|
| Länge (Basen) | 2 | 3 | 4 | 5 |
| HPLC-Reinigung | ja | ja | ja | ja |
| Maldi-Qualitätskontrolle | ja | ja | ja | ja |
| Menge (µmol) ca. | 0,2 | 0,2 | 0,2 | 0,2 |
Oligonucleotide bis 200 Basen
| Unser Sondersynthese-Scale CG umfasst: |
|---|
- Unmodifizierte Oligonucleotide zwischen 140 und 200 Basen
- Optimale Synthese dieser Oligonucleotide durch spezielle Zyklen und ausgewählte Chemikalien
- Ständige Überwachung des Syntheseverlaufs durch Tritylmonitoring
- Garantierte Ausbeute von mind. 250 pmol gereinigtem Oligonucleotid
- Inklusive 2-Stufen-Reinigung: Das Oligo wird zunächst mit HPLC und anschließend mit PAGE gereinigt
Mit welcher Ausbeute an vollständigen und richtigen Klonen kann man bei einem so langen Oligonucleotid rechnen? Wir haben die Probe aufs Exempel gemacht:
In Kooperation mit der Universität Ulm haben wir exemplarisch ein von biomers.net synthetisiertes 200mer mittels PCR amplifiziert, kloniert und sequenziert. Von 10 sequenzierten Klonen zeigen 30% exakt die gewünschte Sequenz, bei einem wurde eine Insertion einer Base an Position 22 gefunden, bei einem weiteren die Deletion einer Base an Position 147.
Bei vier weiteren Klonen wurden neben den zu erwartenden Deletionen von ein oder zwei Basen, sowie einer Transition (C->T), die bei derartig langen Oligonucleotiden nicht mehr vollständig separiert werden können, auch einige Deletionen mit mehreren Basen am Stück gefunden (4, 6, 7 und 16 Basen deletiert). Solche Deletionen können während der Synthese eines Oligonucleotids auf Grund der zyklischen Reaktion der Synthese nicht plausibel erklärt werden, wahrscheinlicher ist hier die Ursache in der PCR zu suchen (Hecker et al., 1998, BioTechniques 24:256-260).
In weiteren Analysen zeigte sich mit verschiedenen Oligonucleotiden zwischen 175 und 200 Basen Länge im Schnitt bei drei von fünf Klonen die korrekte Sequenz.
XXL-DNA
Mid Scale DNA-Synthese (XXL)
für Oligonucleotidmengen von 10 bis 500 mg.
| Synthese-Scale | XXL10MG | XXL25MG | XXL50MG | XXL100MG | XXL250MG | XXL500MG |
|---|---|---|---|---|---|---|
| Menge (mg) | > 10 mg | > 25 mg | > 50 mg | > 100 mg | > 250 mg | > 500 mg |
| Menge (OD), ca. | 400 OD | 850 OD | 1700 OD | 3400 OD | 8500 OD | 17000 OD |
| Menge (µmol) 20mer, ca. | 1,8 µmol | 3,5 µmol | 7,5 µmol | 15 µmol | 38 µmol | 77 µmol |
Die angegebenen Mengenwerte sind ungefähre Werte und gelten für ein 20mer DNA-Oligonucleotid gemischter Sequenz.
Alle XXL-Oligonucleotide werden über HPLC-gereinigt und mit Maldi-Massenspektrometrie kontrolliert.
Wobbles
Wobbles
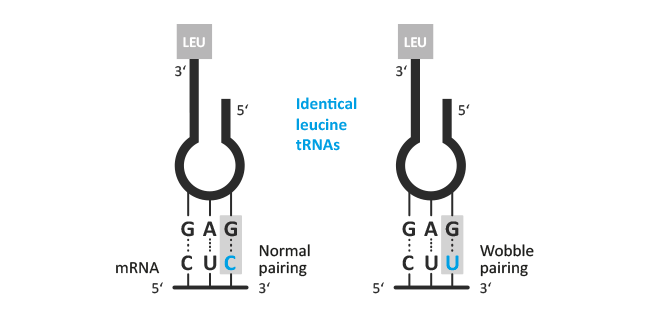
In der Molekulargenetik erklärt die Wobble-Hypothese, wie eine bestimmte tRNA mehr als ein Basentriplett der mRNA (Codon) erkennen kann. Die ersten zwei Positionen des Basentripletts im Codon der mRNA binden üblicherweise die hierzu komplementären Basen des Anticodons der tRNA. Lediglich die erste Position der tRNA ist nicht immer eindeutig festgelegt und lässt z.B. durch Verwendung des seltenen Nucleosids Inosin auch ungewöhnliche Basenpaarungen zu (Wobble-Position).
Mögliche Varianten die sich daraus ergeben sind:
| Guanosin : Uridin | Inosin : Uridin | Inosin : Adenosin | Inosin : Cytidin |
 |
Oligonucleotide mit Wobbles (Mischbasen, degenerierte Basen) finden vielfältige Anwendung. Bereits während der Synthese von Oligonucleotiden können an definierten Stellen Wobbles eingebaut werden. Je nach Art der Mischbase führt das zu bestimmten statistischen Basen-Verteilungen an der jeweiligen Position im Oligo.
Synthese von Wobble-haltigen Oligos:
Viele moderne DNA/RNA-Synthesizer "verstehen" den Einbuchstaben-Code für Wobbles (siehe unten) und mischen die jeweiligen Bausteine während der Synthese automatisch im richtigen Verhältnis (2er-Wobble: 50% der jeweiligen Komponente, 3er-Wobble: jeweils 33% und N=4er-Wobble: jeweils 25%).
Dieses schnelle und einfache Verfahren führt in aller Regel zu guten Ergebnissen mit geringen Abweichungen von den gewünschten statistischen Verteilungen. Abhängig von der jeweiligen Sequenz und des Reaktivitätsunterschieds der Synthesebausteine können die Abweichungen in Einzelfällen auch größer sein.
Vor der Synthese der Oligos kann manuelles Mischen der Basen im richtigen Verhältnis die Ergebnisse in kritischen Fällen oder größeren Syntheseansätzen unter Umständen deutlich verbessern. Des Weiteren sind dadurch auch Mischungen möglich, die von einer Gleichverteilung abweichen.
Kontaktieren Sie hierzu gerne unser Customer Support Team.
Reinigung von Oligos mit Wobbles:
Je nach Art und Anzahl der Wobbles setzt sich das hergestellte Oligonucleotid aus einer Vielzahl von unterschiedlichen Sequenzen zusammen. Diese, unter Umständen mehrere Tausend Sequenzen können sich z.B. in ihrem Laufverhalten in Polyacrylamidgelen (PAGE) oder auf der HPLC-Säule unterscheiden. Bei hochgewobbelten Oligos führt das dann zu einer Verbreiterung der entsprechenden Gelbande bzw. Peaks in der HPLC. Insbesondere beim Ausschneiden von Gelbanden besteht die Gefahr, dass einzelne Sequenzen der Wobble-Mischung „verloren gehen“. Wir empfehlen daher keine PAGE-Aufreinigung von Oligonucleotiden mit Mischbasen, bzw. nur nach Absprache mit unserem Technischen Support Team.
Der nachfolgende, international gültige „Einbuchstaben-Code“ gibt die möglichen Wobble-Kombinationen aus den vier Standardbasen wieder. Bitte verwenden Sie bei der Bestellung von Oligos mit degenerierten Basen diesen Code.
| Symbol | Erklärung | Basen |
|---|
| Normale Basen | |||||
| A | Adenin | A | |||
| C | Cytosin | C | |||
| G | Guanin | G | |||
| T | Thymin | T | |||
| U | Uracil (nur RNA) | U | |||
| Wobble Basen | |||||
| W | Weak (binding bases) | A | T | ||
| S | Strong (binding bases) | C | G | ||
| M | aMino | A | C | ||
| K | Keto | G | T | ||
| R | puRine | A | G | ||
| Y | pYrimidine | C | T | ||
| B | Not A (B comes after A) | C | G | T | |
| D | Not C (D comes after C) | A | G | T | |
| H | Not G (H comes after G) | A | C | T | |
| V | Not T (V comes after T and U) | A | C | G | |
| N | aNy base | A | C | G | T |
Sequenzmodifikationen
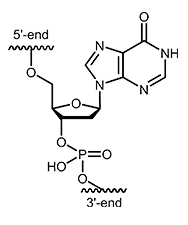
Desoxyinosin (dI)
|
Neben den vier Basen der DNA, Adenin, Cytosin, Guanin und Thymin, findet man in der RNA noch die Basen Uracil und Inosin, sowie weitere seltene Nucleotide. Obwohl Inosin ein Purin ist, kann es sowohl mit Pyrimidinen als auch mit den Purinen der DNA Paarungen eingehen, sodass Inosin auch als „universelle“ oder auch „neutrale“ Base bekannt ist. Manche Paarungen scheinen jedoch energetisch günstiger zu sein als andere, sodass die Bindungen zwischen den einzelnen Basen unterschiedlich stark ausgeprägt sind. Die Bindungsstärke nimmt hierbei über die folgende Reihe ab:1,2 |
 |
| Die folgende Tabelle zeigt die möglichen Fehlpaarungen der Basen an: |
|---|
| tRNA | A | C | G | U | I |
| mRNA | U | G | C | A | A |
| U | G | C | |||
| U | |||||
| G |
Dieser Wobble-Effekt ist auch in Oligonucleotiden möglich. Durch den Einbau eines Inosin-Nucleotids in die Sequenz eines Primers erhält man Primer mit „variablen“ Basen. Diese degenerierten Primer ermöglichen z.B. das Annealing und die Amplifikation mehrerer verwandter Sequenzen.
Bei der PCR mit degenerierten Primern ist allerdings zu beachten, dass proof-reading-Polymerasen, wie beispielsweise Pfu, dazu neigen, von der DNA abzufallen, wenn sie auf Primer mit Wobble-Basen treffen.
Hinweise zur Bestellung:
Bitte wählen Sie bei der Bestellung desoxy- oder ribo-Inosin (2´-dInosin, rI) als interne Modifikation aus. Dazu kennzeichnen Sie die Position der internen Modifikationen in Ihrer Sequenz durch die Zahl (5,6,7,8), mit der das entsprechende Pulldown-Menü im Online-Bestellformular gekennzeichnet ist. Falls Sie keinerlei Modifikationen zur Auswahl angezeigt bekommen, sollten Sie sicherstellen, dass Sie bei Ribobasen weder den Scale "XS" noch die Reinigungsart "Kartusche" ausgewählt haben, da beide nicht mit Modifikationen kombiniert werden können.
Literatur:
1. Studies on the base pairing properties of deoxyinosine by solid phase hybridisation to oligonucleotides. Case-Green SC, Southern EM; Nucleic Acids Res. (1994), 22(2):131-6.
2. Base pairing involving deoxyinosine: implications for probe design. Martin FH, Castro MM, Aboul-ela F, Tinoco I Jr.; Nucleic Acids Res. (1985), 13: 8927-8938.
3. PCR with degenerate primers containing deoxyinosine fails with Pfu DNA Polymerase. Knittel T, Picard D, PCR Methods and Applications (1993), 2: 346-347.
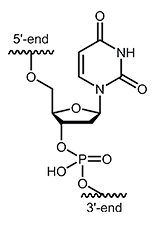
Desoxyuridin (dU)
|
Das Nukleosid Desoxyuridin (dU) besteht aus dem Zucker β-D-Desoxyribose und der Nukleinbase Uracil. Gelegentlich entsteht in lebenden Zellen durch hydrolytische Desaminierung von Cytidin Desoxyuridin. Um eine Falschpaarung während der Replikation der DNA zu verhindern, wird ein Reparaturprogramm in der Zelle eingeleitet. Mithilfe der Uracil-DNA-Glykosylase werden Uracile spezifisch aus der DNA entfernt und abasische Stellen entstehen, die wiederum von Endonukleasen erkannt werden können. Auf diese Weise können gezielt Strangbrüche in der DNA erzeugt werden. Der Einbau von dUTP in einen Primer kann bewirken, dass dieser aus dem Reaktionsmix gezielt entfernt werden kann. Diese Methode findet z.B. bei der Direkt-Sequenzierung von PCR-Produkten Anwendung, bei der ein Primer nach erfolgreicher PCR-Reaktion direkt für die Sequenzierung des PCR-Produktes eingesetzt werden kann. Hinweise zur Bestellung: |
 |
Weitere verfügbare Sequenzmodifikationen finden Sie hier .

