
In situ Hybridisierung
Fluoreszenz in situ Hybridisierung (FISH) ist eine weitverbreitete Methode, die schon seit mehreren Jahrzehnten für viele Fragestellungen eingesetzt wird. Aufgrund der sehr hohen Spezifität und Sensitivität kann FISH auf subzellulärer Ebene Strukturen analysieren oder sogar einzelne Zellen in großen Zellverbänden eindeutig identifizieren und lokalisieren.
Das Nachweisverfahren beruht darauf, fluoreszenzmarkierte Oligonucleotide mit den zelleigenen Nucleinsäuren zu hybridisieren. Die Probe kann sich hierbei gegen DNA oder RNA richten und lässt dabei nicht nur Aussagen über das Vorhandensein eines bestimmten Gens zu, sondern auch das Genprodukt in Form einer RNA - und damit Genaktivität - kann nachgewiesen werden.
Im Bereich der Analyse von Mikroorganismen werden kurze, fluoreszenzmarkierte Oligo-Sonden eingesetzt, die an die zelleigene ribosomale RNA hybridisieren. Die hohe Kopienzahl, in der ribosomale RNA pro Zelle vorliegt, ermöglicht einen äußerst sensitiven Nachweis. Durch die hohe Sensitivität der Fluoreszenz in situ Hybridisierung sind Analysen auch von seltenen und nicht-kultivierbaren Proben möglich, sodass ein weites Feld von Analysen denkbar ist (z.B. marine Mikrobiologie, Analyse mikrobieller Kolonien in Biofilmen oder Biogasanlagen, Mikrobiom-Analysen, etc.).
Neben der subzellulären Auflösung von RNA in Zellen ist auch eine quantitative Messung von RNA in Zellen über die in situ Hybridisierung möglich.
Fluorescent Probes
Fluoreszenz-Sonden für FISH Analysen
Für fluoreszenzmarkierte Oligonucleotid-Proben können stabile und lichtstarke Farbstoffe aus dem gesamten Spektrum des sichtbaren Lichts eingesetzt werden (siehe Fluoreszenzfarbstoffe). Damit können auch bei autofluoreszierenden Umgebungen oder für Multiplex-Analysen passende Kombinationen gefunden werden.
Je nach Fragestellung stehen unterschiedliche Markierungsgrade zur Verfügung:
monoProbes
monoProbes stellen den Klassiker unter den FISH Sonden dar. Hierbei werden die Probes mit einem Fluoreszenzfarbstoff am 5´-Ende des Oligonucleotids markiert.

dopeProbes
Durch zweifache Fluoreszenzmarkierungen der Oligonucleotidsonden kann in FISH Experimenten (DOPE-FISH) eine annähernde Verdopplung der Signalintensitäten und somit höhere Lichtausbeuten pro hybridisierter Probe erreicht werden. Aufgrund der zusätzlichen Sensitivität kann diese Technik auch bei seltenen Targets angewendet werden kann.
Die dopeProbes werden hierbei am 5´- und 3´-Ende mit identischen Fluoreszenzfarbstoffen markiert.

tetraProbes
Eine Weiterentwicklung davon sind tetraProbes, die bis zu vier Farbstoffmoleküle an einem Oligonucleotid tragen und dadurch höchst sensitiv sind. Die Fluoreszenzfarbstoffe werden dabei verteilt über die Oligolänge im Rückgrat angeordnet. In hybridisiertem Zustand verhält sich die Signalintensität nahezu linear zur Anzahl der Farbstoffe pro Oligo, d.h. unter ansonsten identischen Bedingungen leuchten tetraProbes etwa doppelt so hell wie dopeProbes und viermal so hell wie monoProbes.
Neben der hierbei üblichen Anbindung der Fluorophore an die 2´-Gruppe des Zuckers, führt auch die Clickreaktion über die Base 5-Ethynyl-dU zu einer nahezu direkten Modifikation am Oligorückgrat.

multiProbes
Die interne Click-Modifikation findet nicht nur bei der Herstellung von tetraProbes Anwendung. Vielmehr eröffnen sie vielfältige Möglichkeiten Oligonucleotide mehrfach mit identischen Biomolekülen zu markieren.
Bei diesen sogenannten multiProbes sind beispielsweise Oligos mit neun internen Fluoreszenzfarbstoffen denkbar.
Literatur:
1. A Straightforward DOPE (Double Labeling of Oligonucleotide Probes)-FISH (Fluorescence In Situ Hybridization) Method for Simultaneous Multicolor Detection of Six Microbial Populations. Behnama F, Vilcinskasb A, Wagnera M, Stoeckerb K; Appl. Environ. Microbiol. (2012), vol. 78 no. 15 5138-5142.
HRP
HRP-markierte Oligonucleotide
Eine Sensitivitätssteigerung um mehrere Größenordnungen wird durch die Verwendung von HRP-Probes erreicht. Hier wird statt eines Farbstoffs das Enzym Meerrettich-Peroxidase (HRP = Horseradish peroxidase) an die Oligonucleotid-Sonde gekoppelt. Das etwa 40 kDa schwere Enzym setzt an der Stelle, an der das Oligo in der Zelle an seine Zielsequenz bindet, zugegebene Farbstoffe um und führt dadurch am Bindungsort zu einer Amplifikation des Lichtsignals. Durch Kombination von HRP-Probes und Tyramid Signalamplifikations-Systemen (TSA) können, im Vergleich zu Fluorescein-einfach-markierten Sonden, vielfach höhere Signalintensitäten erzielt werden. Diese Methode wird als "Catalyzed Reporter Deposition - Fluorescent in situ Hybridisation" (CARD-FISH) bezeichnet und wird zum quantitativen Nachweis von Mikroorganismen eingesetzt.
Bei der Synthese wird das Oligonucleotid, üblicherweise etwa 18-25 Nucleotide lang, am 5’-Ende mit einem Aminolink modifiziert. Diese reaktive Aminogruppe wird mit einem bifunktionalen Crosslinker gekoppelt und aktiviert. Die zweite reaktive Seite des Crosslinkers wird in einem weiteren Schritt mit einer freien Aminogruppe der Peroxidase zur Reaktion gebracht. Es resultiert ein Konjugat, verknüpft über chemisch stabile kovalente Bindungen. Durch geeignete Wahl der Reaktionsbedingungen und Reagenzienüberschüsse wird in etwa eine 1:1 Kopplung zwischen Oligonucleotid und Peroxidase erreicht.
Zur Reinigung und Isolierung des Konjugates hat sich die Polyacrylamidgelelektrophorese (PAGE) als am besten geeignetes Verfahren erwiesen.
Click-FISH
Click-markierte Oligonucleotide
Der Einsatz alternativer Kopplungsstrategien eröffnet immer vielfältigere und flexiblere Möglichkeiten Modifikationen in eine Oligosequenz einzubringen.
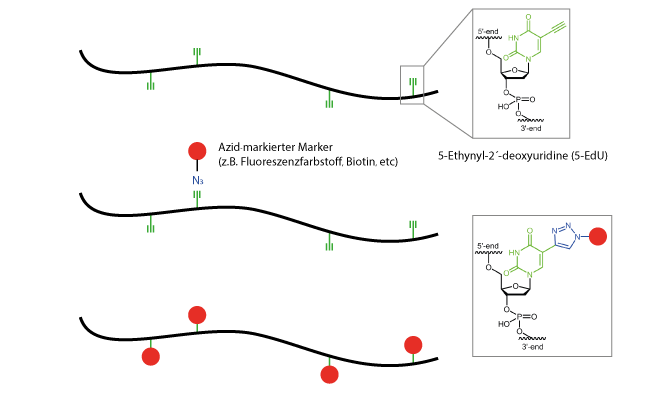
Hervorzuheben ist hierbei die Technologie der internen Click-Modifikation von Nucleinsäuren, der sogenannten Click-FISH Methode. Die Kopplung erfolgt dabei über die heterozyklischen Pyrimidinbasen 5-Octinyl-dC, 5-Octinyl-dU oder 5-Ethinyl-dU. Die modifizierten Basen sind Cytidin und Thymidin Nucleosid-Analoga und werden während der Synthese an entsprechender Position in die wachsende Oligosequenz eingebracht.
Die eigentliche Anbindung des Markermoleküls (z.B. Fluoreszenzfarbstoffe) erfolgt indem die Azid-markierten Marker über die kupferkatalysierte Click-Reaktion mit dem Alkin der modifizierten, Click-fähigen Pyrimidine reagieren. Die Farbstoffe können vor oder auch erst nach der Hybridisierung der Sonde an das Targetmolekül binden.
Diese Form der internen Kopplung kann auch beispielsweise für mehrfach fluoreszenzmarkierte Oligosonden, wie den tetraProbes, angewandt werden.
smiFISH
smiFISH zur in situ RNA Detektion
Bei Genexpressionsanalysen in Zellen und Geweben nimmt die in situ RNA-Detektion einen immer größeren Stellenwert ein. Für den sensitiven Nachweis bestimmter RNA-Moleküle wurden modifizierte FISH-Anwendungen entwickelt, mit denen der Nachweis einzelner Moleküle möglich ist.
Beim klassischen single molecule FISH (smFISH) werden viele kurze Oligonucleotide (typischerweise 24-48 Oligosonden) spezifisch gegen die RNA-Targetsequenz designt, sodass diese nebeneinander auf der Zielsequenz hybridisieren können.
Diese große Anzahl von Oligos, der sogenannte Oligopool, führt für jede RNA zu einem hochspezifischen Signal, das unter einem Standard-Weitfeldmikroskop als einzelner Punkt sichtbar wird.
Im Vergleich zum akkumulierten starken Signal, das von einer großen Anzahl von Oligonucleotiden an der Ziel-RNA generiert wird, ist das Hintergrundsignal einzelner Oligonucleotide normalerweise wesentlich schwächer.
In den ursprünglichen smFISH-Ansätzen wurden die Oligonucleotide des Oligopools direkt markiert. Eine kürzlich vorgestellte Methode namens smiFISH (single molecule inexpensive fluorescence in situ hybridisation) bietet neben der größeren Flexibilität auch eine enorme Kostenersparnis, indem unmarkierte RNA-spezifische Primärsonden verwendet werden.
Jede Primärsonde setzt sich aus einem genspezifischen, Target-bindenden Teil (ca. 26-32 Basen Länge) und der sogenannten FLAP-spezifischen Bindestelle (28 Basen Länge) zusammen. Alle Primärsonden tragen die identische FLAP-Sequenz, sodass eine fluoreszenzmarkierte Sekundärsonde (FLAP) daran binden kann und somit die Detektion der intrazellulären mRNA ermöglicht.
Die Sekundärsonde FLAP ist ein doppelt-markiertes Oligo, das sowohl am 5´- als auch am 3´-Ende einen Fluoreszenzfarbstoff trägt.
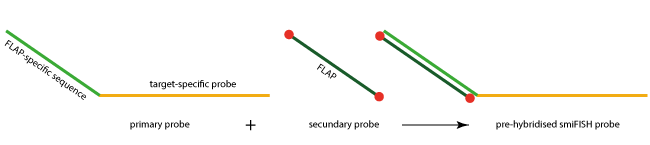
Primäre und sekundäre Sonden können „vorhybridisiert“ und der resultierende Duplex für den RNA-Nachweis verwendet werden. Ein wesentlicher Vorteil dieses smiFISH-Ansatzes ist seine enorme Flexibilität. Primärsonden können dabei leicht mit Sekundärsonden markiert werden, die verschiedene Fluoreszenzfarbstoffe tragen. Auf diese Weise kann ein Set von Sekundärsonden zur Markierung verschiedener Primärsonden verwendet werden.
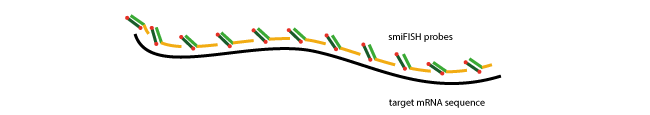
Darüber hinaus ermöglicht die Vorhybridisierung der Primär- und Sekundärsonden die gleichzeitige Visualisierung mehrerer Zielgene - selbst wenn das Set an Primärsonden dieselbe FLAP-Sequenz tragen. Jede RNA-Spezies hybridisiert dabei mit einer Sekundärsonde mit einer unterschiedlichen Farbe. Diese markierten Sondensets können im Anschluss verwendet werden, um diese RNA-Targets in derselben Zelle sichtbar zu machen (Multi-colour imaging assays).
Auf diese Weise konnte smiFISH bereits erfolgreich in verschiedenen kultivierten Zellen, Embryonen und Geweben eingesetzt werden.
Zur Detektion bestimmter RNA in einer Zelle bietet biomers.net standardmäßig drei verschiedene doppelt-markierte FLAP-Sonden an (FLAP X, FLAP Y oder FLAP Z). Diese Sonden können mit einer Vielzahl an möglichen Fluoreszenzfarbstoffen am 5´- und 3´-Ende markiert werden (z.B. Fam, Cyanine 3, Cyanine 5, etc.).
Viele weitere mögliche Fluoreszenzfarbstoffe finden Sie hier.
| Name | FLAP-Sondensequenz (5´-3´) | Länge |
|---|---|---|
| FLAP X | CAC TGA GTC CAG CTC GAA ACT TAG GAG G | 28 nt |
| FLAP Y | AAT GCA TGT CGA CGA GGT CCG AGT GTA A | 28 nt |
| FLAP Z | CTT ATA GGG CAT GGA TGC TAG AAG CTG G | 28 nt |
Das Set der hierzu passenden unmarkierten, Target-bindenden Primärsonden umfasst 24 bzw. 48 unmarkierte Oligonucleotide. Am 3´-Ende jeder Primärsonde wird die jeweils passende FLAP-spezifische Sequenz angefügt.
| Name | FLAP-spezifische Sequenz (angefügt an das 3´-Ende der Primärsonde) (5´-3´) | Länge |
|---|---|---|
| Probe X | CCT CCT AAG TTT CGA GCT GGA CTC AGT G | 28 nt |
| Probe Y | TTA CAC TCG GAC CTC GTC GAC ATG CAT T | 28 nt |
| Probe Z | CCA GCT TCT AGC ATC CAT GCC CTA TAA G | 28 nt |
Unterstrichen ist jeweils ist FLAP-spezifische Sequenz; die „x“ am 5´-Ende stellen die Target-spezifische Sequenz der Primärsonden dar:
Probe X: 5´-xxxxxxxxxxxxxxxxxxxxxxxxxxxxxxCCT CCT AAG TTT CGA GCT GGA CTC AGT G-3´
Probe Y: 5´-xxxxxxxxxxxxxxxxxxxxxxxxxxxxxxTTA CAC TCG GAC CTC GTC GAC ATG CAT T-3´
Probe Z: 5´-xxxxxxxxxxxxxxxxxxxxxxxxxxxxxxCCA GCT TCT AGC ATC CAT GCC CTA TAA G-3´
Ein hilfreiches Protokoll für das smiFISH mRNA-Nachweisassay können Sie im Supplementary Material der Publikation von Tsanov et al. (p. 12-14) finden. Die Detektion der RNA erfolgt mithilfe eines Standard-Weitfeldmikroskops.
Ein äußerst nützliches Tool zur Auswertung der umfangreichen Bildanalyse-Daten stellt die Software FISH-quant dar. Mithilfe des MATLAB-basierten freien Analyseprogramms FISH-quant kann intrazelluläre mRNA detektiert und genau lokalisiert werden.
Die Software wurde am Institut Pasteur von Dr. Florian Müller und Dr. Aubin Samacoits basierend auf Arbeiten von Hervé Marie-Nelly erstellt und erlaubt eine einfache und effiziente Quantifizierung der mRNA bei Einzelmolekül-FISH-Anwendungen (smiFISH, smFISH).
Weitere Informationen zu FISH-quant, einem Link zum Software-Download sowie ausführliche Anleitungen und Testdaten zu smiFISH finden Sie unter https://bitbucket.org/muellerflorian/fish_quant
Besonderer Dank geht an Dr. Florian Müller vom Institut Pasteur, Paris für die hervorragende Zusammenarbeit und die hilfreichen Anregungen und Informationen.
Für weitere Informationen kontaktieren Sie bitte jederzeit gerne unser Kunden Support Team.
Wir beraten Sie gerne bei der Auswahl der für Sie passenden Produkte.
Tel +49 731 70 396 0 I info@biomers.net
Literatur:
1. smiFISH and FISH-quant – a flexible single RNA detection approach with super-resolution capability. Tsanov N, Samacoits A, Chouaib R, Traboulsi A-M, Gostan T, Weber C, Zimmer C, Zibara K, Walter T, Peter M, Bertrand E, Mueller F; Nucleic Acids Research (2016), Vol. 44, No. 22 e165, doi: 10.1093/nar/gkw784.
2. FISH-quant: automatic counting of transcripts in 3D FISH images. Mueller F, Senecal A, Tantale K, Marie-Nelly H, Ly N, Collin O, Basyuk E, Bertrand E, Darzacq X, Zimmer C; Nat Methods. (2013), 10(4):277-8; doi: 10.1038/nmeth.2406.
PNA Probes
PNA Sonden
Besonders im Hinblick auf eine verlässliche Unterscheidbarkeit eng verwandter Spezies – beispielsweise in der Erreger-Diagnostik – sind PNA-Sonden „normalen“ Oligos durch die erhöhte Bindungsspezifität bei gleicher Länge deutlich überlegen. PNA sind durch das Peptid-Rückgrat, das sie von natürlichen Nucleinsäuren unterscheidet, extrem resistent gegenüber Nucleaseverdau und weisen zudem eine höhere Bindungsaffinität auf, sodass auch sehr kurze PNA-Oligonucleotide eine gute Spezifität garantieren. PNA-Sonden sind für Routine-Untersuchungen über lange Zeiträume deshalb gut geeignet.
Links
Links für Probe Design
Für das Design und die Anwendung von Probes im Bereich der in-situ Hybridisierung empfehlen wir Ihnen die folgenden Links:
Arb-Silva (www.arb-silva.de)
Die SILVA rRNA Datenbank ist ein Projekt des Max-Planck-Instituts für Marine Mikrobiologie in Bremen. Die SILVA Datenbank ist die weltweit größte und aktuellste Sammlung ribosomaler RNA-Sequenzen aller Lebensformen (Bakterien, Archaeen und Eukaryoten) und damit für taxonomische und phylogenetische Fragestellungen eine der ersten Adressen. Neben speziell entwickelter Software für das Aligning, Handling und die Auswertung der Sequenzdaten bietet die Webseite eine umfassende Arbeits- und Troubleshooting-Sammlung für Fluoreszenz in situ-Hybridisierungen.
Ribocon (www.ribocon.com)
Ribocon bietet Industrie und Akademien aus dem Bereich der Umwelt-, klinischen und molekularen Mikrobiologie bioinformatische Dienstleistungen und Lösungsvorschläge an. Insbesondere bei mikrobiellen Diversitätsanalysen, phylogenetischen Untersuchungen und beim Design kundenspezifischer Probes verfügt das Ribocon Team über exzellente und jahrelange Erfahrungen.
probeBase (www.microbial-ecology.net/probebase)
Der Onlineserver probeBase wird von der Abteilung der mikrobiellen Ökologie der Universität Wien zur Verfügung gestellt. Er ermöglicht die gezielte Suche von FISH-und Microarray-Sonden und PCR-Primern nach Name, Sequenz oder Zielorganismus. Darüber hinaus ist eine ausführliche Dokumentation der gelisteten Probes verfügbar.
Protocols (www.environmental-microbiology.de/lab_issues.html)
Eine Sammlung informativer Laborprotokolle und hilfreicher Links stellt Ihnen die Abteilung der Mikrobiologie der Technischen Universität München zur Verfügung.
Literatur:
1. Single-molecule fluorescence in situ hybridization: quantitative imaging of single RNA molecules. Kwon S; BMB Rep. (2013),46(2):65-72.
2. Fluorescence In situ Hybridization: Cell-Based Genetic Diagnostic and Research Applications. Cui C, Shu W, Li P; Front Cell Dev Biol. (2016); 4: 89.
3. Applications of fluorescence in situ hybridization (FISH) in detecting genetic aberrations of medical significance. Bishop R; Bioscience Horizons (2010), Volume 3, Issue 1, Pages 85–95.

