
Real-Time PCR Probes
Unter Real-time PCR versteht man eine PCR, die durch die Verwendung geeigneter Hilfsmittel in ihrem Fortgang beobachtet werden kann. Im Gegensatz zu einer herkömmlichen PCR, bei der das Endergebnis im allgemeinen auf einem Agarose-Gel analysiert wird, kann bei der Real-time PCR in jedem einzelnen Zyklus die Zunahme der DNA-Menge -eben in Echtzeit- beobachtet werden. Damit ist eine Quantifizierung möglich, die in Diagnostik und Validierung zu ganz neuen Möglichkeiten geführt hat. Die direkte Beobachtung wird durch die Verwendung fluoreszierender Farbstoffe erreicht. Diese können, wie SybrGreen® und ähnliche Produkte, einfach an doppelsträngige DNA anlagern und so die Generierung neuer Doppelstränge in jedem Zyklus anzeigen.
Eleganter und meist sensitiver, aber auch aufwändiger und nicht für jede Fragestellung geeignet ist die Verwendung von sogenannten Sonden oder 'probes', die zusätzlich zu den Primern in die Reaktion gegeben werden und an das Amplikon binden. Die Bindung der Sonden auf dem amplifizierten Bereich gewährleistet zum einen eine höhere Spezifität, zum anderen kann die Bindung selbst durch die Verwendung geeigneter Hilfsmittel direkt sichtbar gemacht werden. Hierzu werden üblicherweise Fluoreszenzfarbstoffe eingesetzt, deren emittiertes Licht direkt nachgewiesen werden kann.
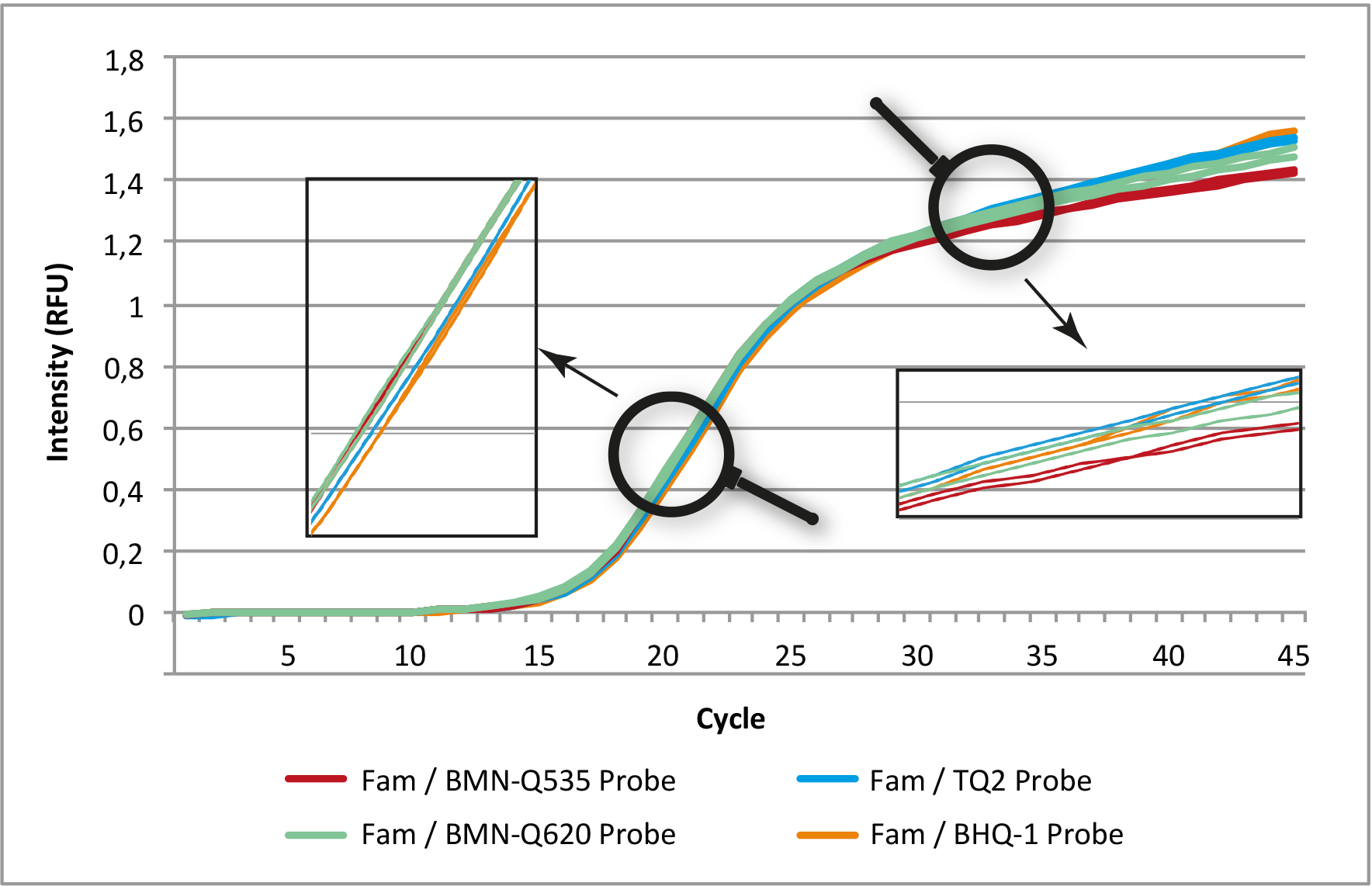
Abbildung 1: Vergleich der Amplifikationskurven (normalisiert) eines qPCR-Assays mit 5´-6-Fam-markierten Sonden und vier verschiedenen 3´-Quenchern.
In jedem Ansatz wurden die gleichen Primer und die gleiche Sondensequenz eingesetzt. Die Assays wurden in zweifacher Ausführung durchgeführt
(Genaxxon ProbeMasterMix No ROX, MyGo Pro, vorkalibrierter Fam-Kanal).
Im direkten Vergleich zeigen die Quencher (BMN-Q535, BMN-Q620, BHQ-1 und TQ2) nahezu identische Ergebnisse mit dem Fluoreszenzfarbstoff 6-Fam.
Mit Hilfe von Quenchern ( von quench=löschen, dämpfen, ersticken) kann die Fluoreszenz gezielt "angeschaltet" werden, sobald die Sonde spezifisch gebunden hat. Grundsätzlich können neben vielen Varianten fünf Grundtypen unterschieden werden, die im folgenden in ihren wesentlichen Punkten kurz dargestellt sind.
DL-Probes - BMN-Q
Dual-Labelled Probes mit BMN-Q460, BMN-Q535, BMN-Q620, BMN-Q1 und BMN-Q2
Bei dem sogenannten TaqMan®-Assay wird eine Sonde so auf der Ziel-DNA plaziert, das diese "im Weg" der Polymerase sitzt.
Im hybridisierten Zustand der Sonde reicht die räumliche Nähe von Fluorophor und Quencher, um die Fluoreszenz zu quenchen (abzuschalten). Während der PCR wird nun die 3´-5´-Exonuklease-Aktivität der Taq-Polymerase genutzt, um die Sonde abzubauen. Dadurch werden Fluorophore und Quencher voneinander getrennt und die Fluoreszenz freigesetzt.
Gerne stehen wir Ihnen bei Fragen zum Design dieser Sonden und den zugehörigen Primern zur Seite.
Dual-Labelled Probes mit BMN-Quenchern
| 5'-Fluorophor | ABS [nm] | EM [nm] | empfohlener BMN-Quencher |
|---|---|---|---|
| Atto 425 | 436 | 484 | BMN-Q460 |
| Atto 465 | 453 | 508 | BMN-Q460 |
| Fam | 494 | 520 | BMN-Q535, BMN-Q1 |
| Joe | 520 | 548 | BMN-Q535, BMN-Q1 |
| Hex | 535 | 556 | BMN-Q535, BMN-Q1 |
| Cyanine 3 | 550 | 570 | BMN-Q620, BMN-Q1 |
| Atto 550 | 554 | 579 | BMN-Q620, BMN-Q1 |
| Atto Rho 11 | 572 | 595 | BMN-Q620, BMN-Q2 |
| Rox | 574 | 602 | BMN-Q620, BMN-Q2 |
| Cyanine 3.5 | 588 | 604 | BMN-Q620, BMN-Q2 |
| Atto Rho 101 | 586 | 610 | BMN-Q620, BMN-Q2 |
| Atto 647N | 646 | 664 | BMN-Q620 |
| Cyanine 5 | 649 | 670 | BMN-Q620 |
| Cyanine 5.5 | 675 | 694 | BMN-Q620 |
* Im Preis inbegriffen ist jeweils ein Oligonucleotid bis max. 30 Basen inklusive HPLC-Reinigung und der Maldi-Qualitätskontrolle mit den oben genannten Fluorophor/Quencher-Kombinationen. Der Preis ist nur gültig für elektronische Bestellung über das biomers.net Online-Bestellformular zzgl. ges. MwSt. und ggf. Versandkosten. Weitere Konditionen ergeben sich aus der jeweils gültigen Preisliste. Preisänderungen und Irrtum vorbehalten.
DL-Probes - BHQ, TQ, BBQ, Tamra
Dual-Labelled Probes mit den Quenchern BHQ, TQ, BBQ und Tamra
| 5'-Fluorophor | ABS [nm] | EM [nm] | 3'-Quencher |
|---|---|---|---|
| Atto 425 | 436 | 484 | BHQ-1 BBQ-650 |
| Fam | 494 | 520 | TQ2 BHQ-1 BBQ-650 Tamra |
| Joe | 520 | 548 | TQ2 BHQ-1 |
| Hex | 535 | 556 | TQ2 BHQ-1 BBQ-650 |
| Cyanine 3 | 550 | 570 | TQ3 BBQ-650 BHQ-2 |
| Atto 550 | 554 | 579 | BHQ-2 BBQ-650 |
| Atto Rho 11 | 572 | 595 | BHQ-2 |
| Rox | 574 | 602 | TQ3 BBQ-650 BHQ-2 |
| Cyanine 3.5 | 588 | 604 | TQ3 BBQ-650 BHQ-2 |
| Atto Rho 101 | 586 | 610 | BHQ-2 |
| Atto 647N | 646 | 664 | BHQ-2 BBQ-650 |
| Cyanine 5 | 649 | 670 | BBQ-650 BHQ-2 |
| Cyanine 5.5 | 675 | 694 | BBQ-650 |
* Im Preis inbegriffen ist jeweils ein Oligonucleotid bis max. 30 Basen im Scale S (garantierte Liefermenge >1OD) inklusive HPLC-Reinigung und der Maldi-Qualitätskontrolle mit den oben genannten Fluorophor/Quencher-Kombinationen. Der Preis ist nur gültig für elektronische Bestellung über das biomers.net Online-Bestellformular zzgl. ges. MwSt. und ggf. Versandkosten. Weitere Konditionen ergeben sich aus der jeweils gültigen Preisliste. Preisänderungen und Irrtum vorbehalten.
DQ-Probes
Höhere Sensitivität für Ihre qPCR mit Double Quenched Probes von biomers.net
Die üblichen qPCR Sonden für 5'-Nuclease Assays tragen am 5'-Ende einen Fluorophor und am 3'-Ende einen passenden Quencher, welcher zum einen die Fluoreszenz des Farbstoffes löscht und zum anderen die Verlängerung der Sonde während der qPCR-Reaktion verhindert. Bei längeren Sonden und damit größerem Abstand zwischen Fluorophor und Quencher nimmt der Background oft in ungewünschtem Maße zu.
Mit Double Quenched Probes verringert sich der Background und erhöht sich die Sensitivität. Durch Einbau eines zusätzlichen Quenchers (z.B. BMN-Q535 oder BMN-Q620) in die Sequenz nahe des 5'-Fluorophors kann eine deutliche Verbesserung der Resultate erzielt werden.
Double Quenched Probes enthalten einen 5'-Fluorophor, einen 3'-BMN-Quencher sowie einen internen BMN-Quencher in einem Abstand von 8-10 Basen vom Fluorophor.
| Dual-Labelled Probe | Double Quenched Probe |
|---|---|
|
|
 |
| 5'-Fam, Hex, Cyanine, Atto-Dyes, uvm. 3'-BMN-Q535, BMN-Q620 |
5'-Fam, Hex, Rox, Cyanine, Atto-Dyes, uvm. int. BMN-Q535, BMN-Q620 3'-BMN-Q535, BMN-Q620 |
Standard Dual-Labelled Probes und Double Quenched Probes mit BMN-Quenchern können über das biomers.net Web-Bestellformular online bestellt werden.
Eine ausführliche Bestellanleitung für Double Quenched Probes finden Sie hier.
Für weitere Informationen und Unterstützung bei der Bestellung kontaktieren Sie bitte gerne unseren Kundenservice.
MGB
Minor Groove Binder (MGB)
MGB-modifizierte Oligonucleotide besitzen eine erhöhte Schmelztemperatur (Tm) und Spezifität und können äußerst stabile Duplexe mit komplementären DNA-Sequenzen bilden. Hierbei wird die Schmelztemperatur Tm der modifizierten Oligos stark von der Basenzusammensetzung beeinflusst.
Bei der Bindung der Sonde an die Zielsequenz binden Minor Groove Binder (MGB) an die kleine Furche der DNA. Die sichelförmige Gestalt der MGB CDPI3 (Dihydropyrroloindolecarboxylate Tripeptide CDPI3) erlaubt eine exakte räumliche Anpassung an die gekrümmte Struktur der kleinen Furche und ermöglicht damit eine Einlagerung zwischen die Zucker-Phosphat-Ketten des DNA-Rückgrats. Diese isohelikale Konformation wird dabei vor allem mittels wirkender Van-der-Waals Kräfte und hydrophober Wechselwirkungen stabilisiert.
Auf diese Weise kann eine hohe Stabilität des doppelsträngigen Moleküls erzielt werden, sodass auch sehr kurze DNA-Sonden äußerst effizient an die Target-DNA hybridisieren können.
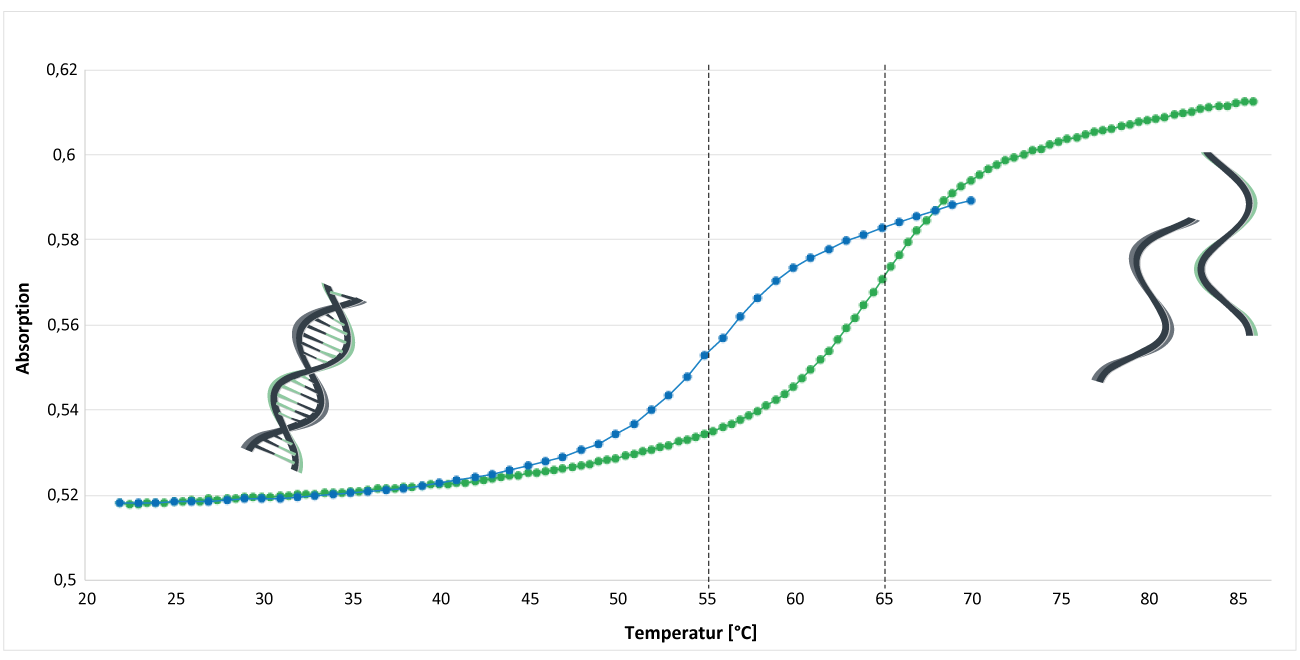
Abbildung 1 zeigt die Schmelzkurvenanalyse eines MGB CDPI3-modifizierten und eines unmodifizierten Oligos. Der Analyse wurde in Phosphatpuffer bei einem pH-Wert von 7.4 durchgeführt. Die Natrium-Konzentration betrug 30 mM; die eingesetzte Konzentration der DNA-Einzelstränge lag bei ca. 1,3 µM. Der Wendepunkt der Schmelzkurve gibt jeweils die Schmelztemperatur Tm des Oligos an.
Die blaue Kurve stellt ein unmodifiziertes EUB338-Oligo dar: 5´-gctgcctcccgtaggagt-3´. Der Schmelzpunkt liegt hier bei ca. 55°C.
Die grüne Kurve zeigt die Schmelzkurve eines MGB CDPI3-modifizierten Oligos. Das EUB-Oligo wurde am 3´-Ende mit einer MGB CDPI3-Modifikation markiert: 5´-gctgcctcccgtaggagt-MGB CDPI3. Der hier ermittelte Schmelzpunkt liegt bei ca. 65°C.
Im direkten Vergleich der beiden Oligonucleotide kann unter den hier gewählten Bedingungen ein Anstieg der Schmelztemperatur um ca. 10°C festgestellt werden.
Eine ausführliche Bestellanleitung für MGB Probes finden Sie hier.
Für weitere Informationen und Unterstützung bei der Bestellung kontaktieren Sie bitte gerne unseren Kundenservice.
Literatur:
1. 3′-Minor groove binder-DNA probes increase sequence specificity at PCR extension temperatures. Kutyavin IV, Afonina IA, Mills A, Gorn VV, Lukhtanov EA, Belousov ES, Singer MJ, Walburger DK, Lokhov SG, Gall AA, Dempcy R, Reed MW, Meyer RB, Hedgpeth J; Nucleic Acids Research (2000), Volume 28, Issue 2, Pages 655–661.
2. Minor Groove Binder-Conjugated DNA Probes for Quantitative DNA Detection by Hybridization-Triggered Fluorescence. Afonina IA, ReedMW, Lusby E, Shishkina IG, Belousov YS; BioTechniques (2002) 32:940-949 .
Beacons
Molecular Beacons
Eine weitere Variante stellen die Molecular Beacons® dar. Wie bei den daraus abgeleiteten Scorpions®-Oligos, weisen Molecular Beacons eine ´stem´ -Region (stem=Stamm) auf. Diese setzt sich aus einer kurzen Region zueinander komplementärer Nucleinsäuren zusammen, sodass sich das Molekül mit sich selbst falten kann. Die spezifische Sondensequenz, die komplementär zur Targetsequenz ist, bildet in dieser Konformation die sogenannte Loop-Region.
In Abwesenheit eines passenden Targets werden die jeweils terminal gekoppelten Modifikationen (Farbstoff und Quencher) durch den ´stem´ in enger räumlicher Nähe gehalten, sodass die Fluoreszenz inhibiert wird. In diesem Zustand überträgt der Fluorophor seine Elektronen auf den Quencher, der diese Energie wiederum in Form von längerwelligen Licht oder Wärme abgibt.
In Anwesenheit eines passenden Targetmoleküls öffnet sich der Beacon durch Bindung der ´loop´-Region an die entsprechende Zielsequenz. Dieses Sonden-Target-Hybridmolekül ist länger und so konzipiert, dass die Bindung an die Targetregion thermodynamisch begünstigt ist und deshalb letztendlich zur Öffnung des Molecular Beacons führt. Die Änderung der Konformation trennt hierbei Fluorophor und Quencher, sodass die Fluoreszenz freigesetzt werden kann.
Molecular Beacons sind äußerst spezifisch und sind im Idealfall in der Lage Unterschiede zu identifizieren, die lediglich auf einem einzelnen Nucleotid beruhen. Die Länge der Sondensequenz wird dafür so gewählt, dass sie ein perfekt komplementäres Sonden-Target-Hybridmolekül bilden können, das gerade stabiler ist als die komplementäre ´stem´-Region. Bereits ein abweichendes Nucleotid in der Sondensequenz verringert dann die Stabilität des Sonden-Target-Moleküls ausreichend, sodass die Stabilität der ´stem´-Region überwiegt und die Öffnung des Molecular Beacon verhindert wird.
Aufgrund ihres besonderen Verhaltens sind Molecular Beacons vielseitig einsetzbar:
- PCR
- Detektion bestimmter RNAs in lebenden Zellen1
- Amplicon-Detektion in der Diagnostik
- Multiplex Assays zur Detektion verschiedener Targets in einem Reaktionsansatz
- SNP Detektion
- Pharmakogenetische Anwendungen
- Genetisches Screening (Genotypbestimmung eines Locus)
Unter www.molecular-beacons.org erhalten Sie umfassende Informationen über Molecular Beacons und ihre Einsatzgebiete.
Wir freuen uns, Ihnen den Service unseres Probe Designs für Molecular Beacons®, Scorpions® und TaqMan® Probes anbieten zu können.
Schicken Sie uns hierzu einfach die Basensequenz Ihres Targets, das Sie testen möchten. Unsere erfahrenen Spezialisten designen Ihre individuelle Probe.
Wenn Sie weitere Informationen zu diesem Thema wünschen, kontaktieren Sie uns bitte jederzeit.
Tel +49 731 70 396 0 ǀ info@biomers.net
| 5‘-Fluorophor | 3‘-Quencher |
|---|---|
| 6-Fam, Hex, Tet | BMN-Q535, Dabcyl, DDQ-l, BHQ-1, TQ-2, BBQ-650 |
| Yakima Yellow | BHQ-1, TQ-2, BBQ-650 |
| Cyanine 3, Cyanine 5, Tamra, Rox, TexasRed |
BMN-Q620, BHQ-2, TQ-3, BBQ-650 |
| Atto dye, DY dye | BMN-Q, BHQ, TQ, BBQ-650 |
Preise entnehmen Sie bitte der aktuellen Preisliste. Weitere Kombinationen auf Anfrage.
Literatur:
1. Real time detection of DNA.RNA hybridization in living cells. Sokol DL, Zhang X, Lu P, Gewirtz AM; Proc Natl Acad Sci USA (1998),11538-43.
Scorpions
Scorpions
Scorpions-Primer sind mehrfach modifizierte Spezialanfertigungen, die bei einer besonderen Variante der Real-time PCR für hochspezifische Signale eingesetzt werden.
Scorpions-Oligos tragen am 5´-Ende eine Fluorophore, die bei unhybridisierten Sonden durch eine entsprechend designte 'stem'-Struktur' in enger räumlicher Nähe zu einem Quencher gehalten wird. In 3´-Richtung folgt dem Quencher ein Primer-Bereich, der in der PCR-Reaktion zusammen mit dem unmodifizierten Gegenprimer zur Amplifikation der gewünschten Ziel-DNA führt. Um das Auffüllen der Scorpions-Sonde zum Doppelstrang bei der PCR zu verhindern, wird eine weitere Modifikation verwendet, ein sogenannter Blocker, der die Polymerase daran hindert, den 5´-gelegenen Bereich des Scorpions-Oligos zu erreichen.
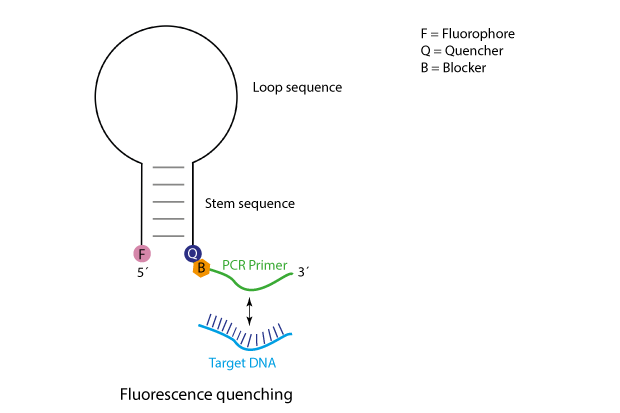
In der PCR-Reaktion ist der Ablauf der folgende:
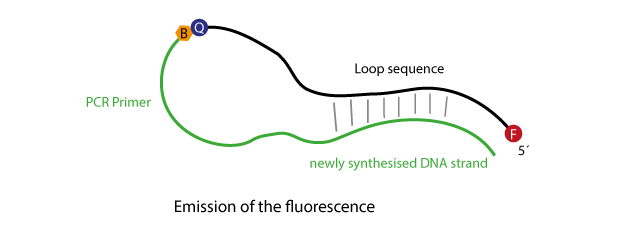
Zunächst lagert sich der Primer-Bereich an die entsprechende Zielsequenz an, nach der Elongation des Scorpions-Primers kann sich die 'stem'-Struktur öffnen und mit dem neu synthetisierten Strang hybridisieren.
Dadurch wird die Fluorophore aus dem Einflussbereich des Quenchers entfernt, so dass die Fluoreszenz nun zur Detektion genutzt werden kann.
biomers.net bietet Scorpions mit folgenden Konditionen an:
| 5‘-Fluorophor | Quencher | Blocker |
|---|---|---|
| 6-Fam, Hex, Tet | int. BHQ-1, int. Dabcyl | Spacer18 |
| Cyanine 3, Cyanine 5 | int. BHQ-2, int. Dabcyl | Spacer18 |
| Atto dye, DY dye | int. BHQ | Spacer18 |
Preise entnehmen Sie bitte der aktuellen Preisliste. Weitere Kombinationen auf Anfrage.
Scorpions Primer werden mit Lizenz von DxS hergestellt.
Preise sind inklusive DNA-Oligonucleotid bis 60 Basen, 5´-Fluorophore, interner Kopplung des Quenchers sowie des Polymerase-Blockers, HPLC-Reinigung und Maldi-Qualitätskontrolle und gelten nur für elektronische Bestellungen.
Fret Probes
Fret Probes
Eine verwandte Anwendung findet sich im LightCycler®-Assay. Dazu werden zwei Sonden hintereinander auf der Ziel-DNA plaziert. Durch die Bindung beider Sonden wird eine extrem hohe Spezifität der Real-time PCR erreicht, die zum Beispiel eng verwandte Gene oder Organismen differenzieren kann.
Die 5´-gelegene Sonde trägt an ihrem 3´-Ende eine Fluorophore, deren Fluoreszenz nicht direkt gemessen, sondern zur Anregung der Fluorophore der zweiten Sonde genutzt wird. Durch diese Übertragung (FRET: fluorecsence resonance energy transfer) wird bei erfolgreicher Bindung beider Sonden die Fluoreszenz der 3´-gelegenen Sonde detektiert.


