Antisense Oligos
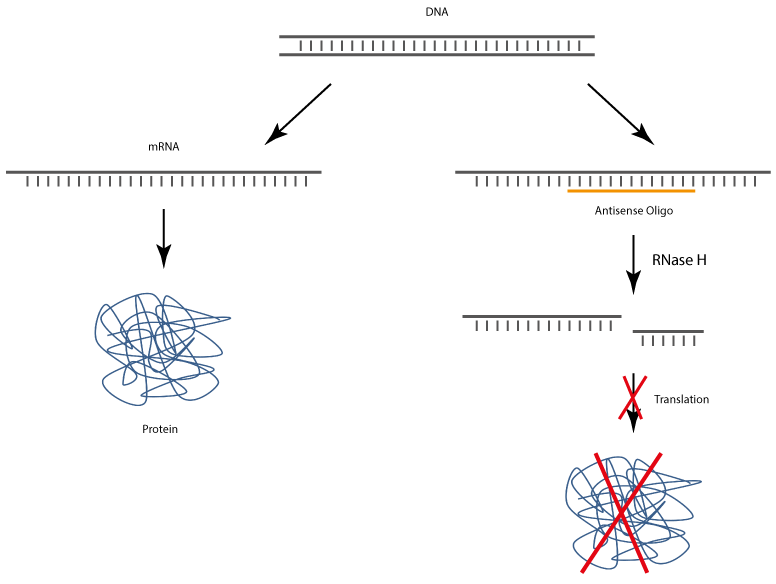
Antisense-Oligonucleotide binden nach Einbringung in die Zielzellen an entsprechend komplementäre mRNA, wodurch die Translation und somit auch die Biosynthese des Zielproteins spezifisch verhindert wird.
Der Komplex aus Antisense-DNA-Olignucleotid und dem gebundenen RNA-Strang wird von der RNase H erkannnt, die die RNA enzymatisch spaltet, wodurch das Antisense-DNA-Oligonucleotid frei wird und somit erneut zur Bindung komplementärer RNA zur Verfügung steht.
Kritische Aspekte sind zum einen die Einbringung in die Zellen, denn die negativ geladene DNA überwindet nur schlecht Zellmembranen. Verschiedene lipophile Moleküle haben sich hierbei als hilfreich erwiesen.
Zum anderen werden ungeschützte, kurze DNA-Oligonucleotide in Zellen schnell abgebaut. Um den Abbau zu verhindern oder zumindest zu verlangsamen, werden neben Phosphothioaten verschiedene 2´-Modifikationen erfolgreich eingesetzt.
Phosphothioate
Phosphothioat-Oligonucleotide (Phosphorothioat, PTO)
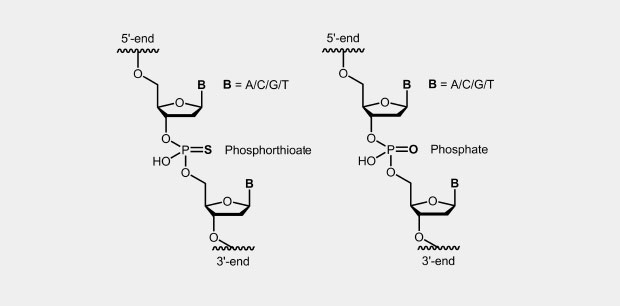
Die Phosphothioat-Modifikation des DNA-Rückgrates ist eine der ersten und auch heute noch vielfach verwendeten Modifikation für Antisense-Oligonucleotide (Antisense Oligos der 1. Generation). Dabei wird im Phosphat-Rückgrat eines der beiden nicht an der Internucleotidbrücke beteiligten Sauerstoffatome durch ein Schwefelatom ersetzt (siehe Abb.). Das zentrale Phosphoratom ist dadurch aufgrund der vier unterschiedlichen Substituenten ein Chiralitätszentrum was Diastereomerenbildung zur Folge hat.
In einem Oligonucleotid können alle Phosphate durch Phosphothioat ersetzt werden (Full-PTO) oder auch nur einzelne (Teil-PTO). Um die gewünschte Stabilität gegen Abbau zu erzielen, scheint es oft ausreichend jeweils an den Enden des Oligos zwei bis vier PTOs einzubauen.
|
|
Bestellinformation:
Phosphothioate werden im Bestellsystem von biomers.net durch Sternchen (*) dargestellt. Sollen alle oder einzelne Phosphodiester durch Phosphothioate ersetzt werden, muss an der jeweiligen Stelle ein * in die Sequenz eingefügt werden.
Beispiele:
- Eine normale Sequenz ohne Phosphothioate:
agcgtgacgtggacgtgagtga - Die identische Sequenz, jedoch als "Full-PTO-Oligo" (alle internucleotidischen Phosphodiester gegen PTO ausgetauscht):
a*g*c*g*t*g*a*c*g*t*g*g*a*c*g*t*g*a*g*t*g*a - Die Sequenz mit einzelnen PTOs ("Teil-PTO"), in diesem Fall jeweils drei PTOs an den Enden:
a*g*c*gtgacgtggacgtgag*t*g*a - Die Sequenz mit einer PTO-Brücke ("Teil-PTO") am 3'-Ende:
agcgtgacgtggacgtgagtg*a
Soll ein PTO-Oligo an einem Ende noch modifiziert werden, beispielsweise mit einem Fluoreszenzfarbstoff, ist es oft sinnvoll auch die Phosphatbrücke zwischen Oligo und Modifikation zu "stabilisieren", also als PTO zu synthetisieren.
Bei einer 5'-Modifikation wird in diesem Fall noch ein * an das 5'-Ende des Oligos angefügt (bei einer 3'-Modifikation entsprechend an das 3'-Ende) und die gewünschte Modifikation im Menü ausgewählt:
- *a*g*c*g*t*g*a*c*g*t*g*g*a*c*g*t*g*a*g*t*g*a, 5'-Modifikation: Biotin : PTO zwischen Biotin und Oligo
2'-modifizierte RNA
2'-modifizierte RNA
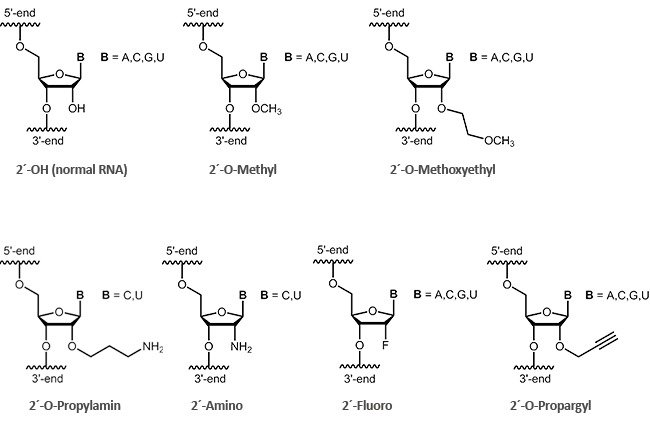
Antisense Oligos der zweiten Generation umfassen RNA-Olignucleotide mit Alkylmodifikationen an der 2´-Position des Ribosezuckers. Die beiden wichtigsten Vertreter dieser Modifikationen sind 2´-O-Methyl-(OMe) und 2´-O-Methoxyethyl-(MOE)-RNAs.
Wenn am 2´-Kohlenstoff des Zuckers ein Sauerstoff verknüpft ist, handelt es sich definitionsgemäß um eine Ribose. Antisense-Oligonucleotide, die eine 2´-Ribose-Modifikation (2´-O-Methyl oder 2´-O-Methoxyethyl) aufweisen, zeigen zumeist in vitro eine erhöhte Stabilität gegen enzymatische Degradation. Hieraus kann auch eine leichte Verbesserung der Verteilung der Oligos in der Zelle abgeleitet werden.1 Des Weiteren konnte beobachtet werden, dass 2´-modifizierte Oligonucleotide (2´-O-Methyl, 2´-O-Methoxyethyl, 2´-O-Propyl, 2´-O-Fluoro) mit größerer Affinität an komplementäre RNA hybridisieren.2
biomers.net bietet Ihnen hierbei eine Auswahl an verschiedenen Modifikationen an:
|
 |
Fragen Sie unseren Kundensupport nach Verfügbarkeit und Lieferzeit.
Tel +49 731 70 396 0 | info@biomers.net
Literatur:
1. Pharmacokinetic Properties of 2´-O-(2-Methoxyethyl)-Modified Oligonucleotide Analogs in Rats. Geary RS, Watanabe TA, Truong L, Freier S, Lesnik EA, Sioufi NB, Samor H, Manoharan M, Levin AA; The Journal of Pharmacology and experimental Therapeutics (2001), Vol. 296, No. 3; JPET 296:890–897.
2. Evaluation of 2´-Modified Oligonucleotides Containing 2’-Deoxy Gaps as Antisense Inhibitors of Gene Expression. Monia BP, Lesnik EA, Gonzalez C, Lima WF, McGee D, Guinosso CJ, Kawasaki AM, Cook PD, Freier SM; The Journal of Biological Chemistry (1993), Inc. Val. 268, No. 19, pp. 14514-14522.
Gapmer Oligos
Gapmer Oligonucleotide
Allgemein sind Antisense Oligos in der Lage die Genexpression zu regulieren, indem der Translationsmechanismus inhibiert und die enzymatische Spaltung der Target-RNA induziert wird.
Die in der Vergangenheit häufig verwendeten Antisense Oligonucleotide der ersten oder zweiten Generation (PTO- oder 2´-modifizierte Oligos) weisen zwar im Gegensatz zu unmodifizierten Oligos eine erhöhte Stabilität im Serum auf, zeigen jedoch im Vergleich zu anderen Antisense Oligonucleotiden toxische Nebeneffekte, die das Anwendungsspektrum dieser Oligonucleotide stark limitieren können.
PTO-modifizierte Antisense Oligonucleotide zeigen zudem geringere Bindungskapazitäten zu komplementären Basen (reduzierte Tm) sowie eine unspezifische Bindung an Proteine.
Antisense Oligos der neueren Generation stellen sogenannte Gapmere dar. Ein Gapmer ist ein einzelsträngiges DNA-Oligo, das spezifisch an endogene Target-mRNA-Sequenzen binden kann. Der entstandene mRNA-Gapmer-Duplex wird von der RNase H erkannt, wodurch der enzymatische Abbau der mRNA induziert und somit die spezifische Expression des Targetgens inhibiert wird.
Ribonucleasen des Typs H sind ubiquitäre Enzyme, die sowohl im Zellkern als auch im Zytoplasma aller Lebewesen zu finden sind. RNase H ist eine sequenzunspezifische Endonuclease, die die RNA von RNA-DNA Heteroduplexen hydrolysiert, nachdem ein Antisense Oligo spezifisch an die Target-mRNA gebunden wurde. Die degradierte mRNA kann auf diese Weise nicht in das entsprechende Protein translatiert werden, was zu einer Down-Regulation des Target Proteins führt (Gene Silencing).
Aufbau Gapmer
Gapmere sind chimäre Oligonucleotide, die aus einem zentralen Block aus DNA-Nucleotiden bestehen, die auf beiden Seiten von modifizierten Basen flankiert sind. Hierbei sind 2´-O-modifizierte Ribonucleotide als auch andere künstlich modifizierte Nucleotidmonomere (z.B. LNA) möglich, die den DNA-Block vor enzymatischen Nucleaseabbau schützen.
Zumeist werden sieben bis acht unmodifizierte DNA-Basen als zentraler Block benötigt, um die RNase H vollständig zu aktivieren und die Spaltung der Ziel-mRNA einzuleiten. An den 3´- und 5´-Enden hat sich gezeigt, dass bereits drei modifizierte Basen ausreichen, um das Oligo gegen Exonucleaseabbau zu schützen und die Halbwertszeit des Oligos um das zehnfache zu erhöhen.
Solche optimal designten Gapmer Oligos erzielen eine höhere Affinität und Sequenzspezifität zum komplementären Gegenstrang und zeigen eine hohe biologische Stabilität bei gleichzeitig geringer Toxizität in biologischen Serum und Zellmedium. So kann beispielsweise bei LNA-Gapmeren die Schmelztemperatur mit jeder eingefügten LNA-Base um 1,5-4°C erhöht und eine höhere Affinität zu komplementären Sequenzen erzielt werden.
Literatur:
- Design of antisense oligonucleotides stabilized by locked nucleic acid. Kurreck J, Wyszko E, Gillen C, Erdmann VA; Nucleic Acids Research (2002), Vol. 30, No.9.
- GapmeR cellular internalization by macropinocytosis induces sequence-specific gene silencing in human primary T-cells. Fazil MHUT, Ong ST, Chalasani MLS, Low JH, Kizhakeyil A, Mamidi A, Lim CFH, Wright GD, Lakshminarayanan R, Kelleher D, Verma NK. Scientific Reports (2016), 6:37721.
Chimären
Chimäre Oligonucleotide
Chimäre Oligonucleotide können aus verschiedenen Arten von Bausteinen hergestellt werden. Im Gegensatz zu natürlich vorkommenden Molekülen können synthetisch hergestellte Oligos gleichzeitig aus DNA und RNA oder PNA bestehen. Ebenso ist es möglich, dass diese Oligos aus rechts- und linksdrehenden Bausteinen gemischt werden (siehe L-DNA, L-RNA, PNA).
Des Weiteren sind Mischungen mit reversen Bausteinen möglich, wodurch Moleküle generiert werden können, die an beiden Enden mit 3´ oder 5´ aufhören.
Gerne stehen wir Ihnen für weitere Auskünfte zur Verfügung.
Kontaktieren Sie uns. Wir helfen Ihnen gerne weiter.
Literatur:
- An Antisense Oligonucleotide Primer. Hogrefe RI; Antisense and Nucleic Acid Drug Development (1999), 9(4): 351-357.
Modifikationen
Antisense Oligonucleotid-Modifikationen
Die wenig zielgerichtete Aufnahme der Oligonucleotidkonjugate in die Zelle stellt häufig die größte Schwierigkeit einer erfolgreichen Entwicklung neuer Oligonucleotid-basierter Mittel dar.
In Bezug auf Antisense-Oligonucleotide ist eine Vielzahl von Modifikationen verfügbar. Abgestimmt auf die jeweilige kundenspezifische Methode sind verschiedene interne bzw. endständige Veränderungen eines Oligonucleotids möglich. Mithilfe kleiner, an das Oligo angehängter Liganden kann die Aufnahme in die Zelle, sowie die zelluläre Verteilung dieser Oligonucleotidkonjugate beeinflusst werden, sodass sie für ein breites Spektrum therapeutischer Anwendungen eingesetzt werden können.
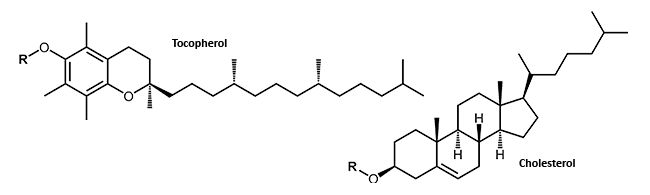
Kleine lipophile Moleküle, die an ein Oligonucleotid angehängt werden, können der stark negativ geladenen DNA oder RNA den Durchgang durch die Zellmembran und somit die Aufnahme in die Zelle erleichtern. So kann beispielsweise das Anhängen von Cholesterol an DNA- oder RNA-Moleküle die Stabilität und die Verteilung der Konjugate in der Zelle verbessern1 (Lipophile Modifikationen).
 |
|
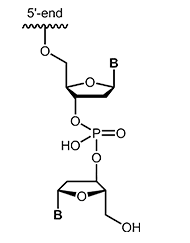
Modifikationen, die ebenfalls den Abbau der Oligonucleotide durch Nukleasen inhibieren können, stellen inverted-ends dar. An das 3´-terminale Nucleotid wird hierbei ein weiteres Nucleotid „verkehrt herum“ angehängt, sodass das Oligo an beiden Enden einen 5´-Terminus besitzt. Diese Art Schutzkappe am 3´-Terminus schützt das Oligonucleotid zum einen vor dem Abbau durch Exonukleasen, gleichzeitig wird die Elongation durch die Polymerase während der PCR gehemmt. |
 |
biomers.net bietet Ihnen in Bezug auf Antisense-Oligos eine umfangreiche Auswahl an Liganden und weiteren Modifikationen an. Unser erfahrenes Support Team hilft Ihnen gerne jederzeit bei allen Aspekten der Auswahl und des Einsatzes Ihrer spezifischen Oligonucleotide weiter.
Literatur:
1. Oligonucleotide conjugates for therapeutic applications. Winkler J; Ther. Deliv. (2013), 4(7),791-809.
Literatur:
- An antisense oligonucleotide primer. Hogrefe RI; Antisense and Nucleic Acid Drug Development (1999), 9(4): 351-357.
- Prospects for nucleic acid-based therapeutics against hepatitis C virus. Lee CH, Kim JH, Lee S-W. World J Gastroenterol. (2013), 19(47): 8949–8962.
- CpG-A and CpG-B oligonucleotides differentially enhance human peptide–specific primary and memory CD8 T-cell responses in vitro. Rothenfusser S, Hornung V, Ayyoub M, Britsch S, Towarowski A, Krug A, Sarris A, Lubenow N, Speiser D, Endres S, Hartmann G; Blood (2004), Vol.103, Number 6.
- CpG-A Oligonucleotides Induce a Monocyte-Derived Dendritic Cell-Like Phenotype That Preferentially Activates CD8 T Cells. Krug A, Rothenfusser S, Selinger S, Bock C, Kerkmann M, Battiany J, Sarris A, Giese T, Speiser D, Endres S, Hartmann G;. J Immunol (2003), 170:3468-3477.