Hapten Modifikationen
Haptene sind kleine Moleküle, die durch spezifische Antikörper erkannt werden. An ein Oligonucleotid gebunden, ermöglicht die Hapten-Modifikation Nachweis, Lokalisierung und Isolierung mit Antikörper-basierten Methoden. Haptene können zur nicht-radioaktiven Markierung von Oligonukleotid-Sonden für die Southern, Northern oder in situ Hybridisierung (ISH) mit anschließender Immundetektion oder für die Nukleinsäure-Reinigung über Affinitätschromatographie verwendet werden. Das Biotin-Streptavidin-System nutzt, in analoger Weise, die spezifische und hoch affine Interaktion zwischen Biotin und Streptavidin.
| Abkürzung | Modifikation | ||||
|---|---|---|---|---|---|
| 5’ | 3’ | 5’ / 3’ doppelt | intern | ||
| Biotin | Bio | x | x | x | x |
| Digoxigenin | DIG | x | x | x | x |
| Dinitrophenol | DNP | x | x | x | |
| Fluorescein | 6-Fam FITC |
x x |
x x |
x x |
x x |
Biotin
Oligonucleotide mit Biotin-Modifikation
Bei biomers.net erhalten Sie verschiedene Biotin-Modifikationen:
Biotin
Die Biotin-Streptavidin-Bindung ist eine der stärksten nicht-kovalenten Bindungen, die in der Natur vorkommen. Sie wird für eine Vielzahl von Methoden und Protokollen genutzt, so daß sich das Biotin-Streptavidin-System in weiten Bereichen der Molekularbiologie, Biochemie und Immunologie etabliert hat. Biotinylierte Oligos finden z. B. als Hybridisierungs-Sonden Verwendung (Southern, Northern, ISH, Microarray). Die Biotin-Markierung wird außerdem zur Isolierung, Abtrennung und Reinigung von DNA oder RNA mittels Biotin-Streptavidin-Affininätschromatographie oder magnetischen Beads genutzt. Spezielle Methoden wie RACE oder Pyrosequencing nutzen die Möglichkeiten der Biotin-basierten Reinigung, indem die zu analysierenden Sequenzen markiert und an Streptavin-aktivierte Oberflächen gebunden werden.
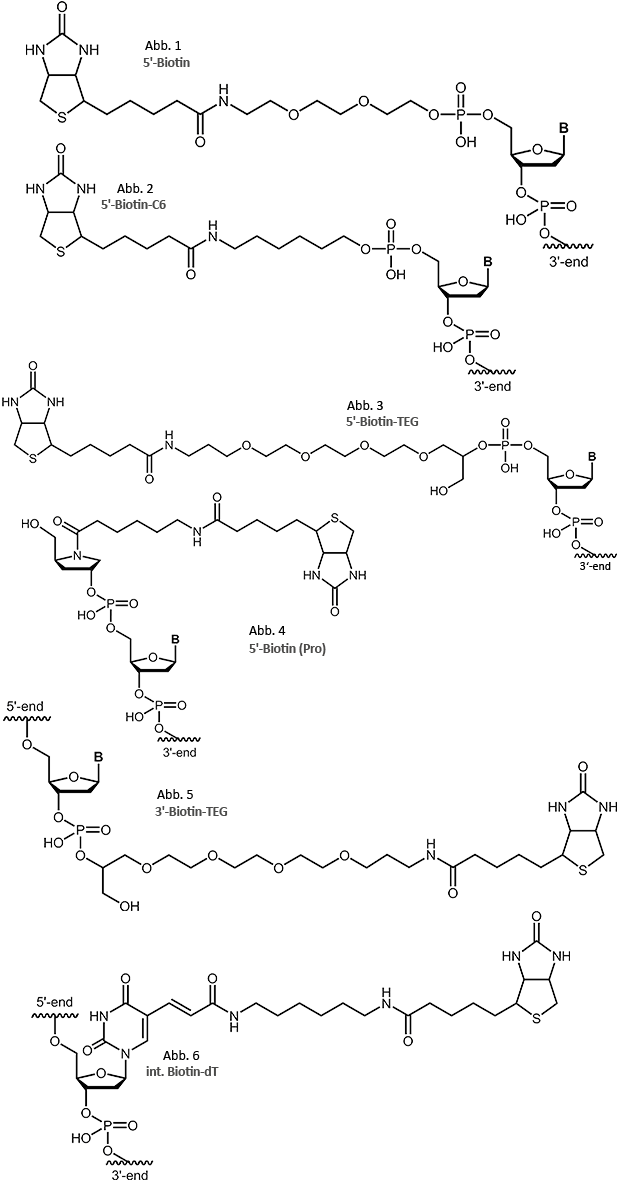
Biotin, das auch als Vitamin H oder Vitamin B7 bekannt ist, kann auf unterschiedliche Weisen und an verschiedenen Stellen an ein Oligo gekoppelt werden. Um verschiedene Abstände zwischen Biotin-tragendem Oligo und dem Streptavidin (insbesondere bei Oberflächen-gebundenem Streptavidin) zu ermöglichen, stehen besonders für Kopplungen am 5'-Ende mehrere Linker zur Verfügung. Neben unserem "Standard-Linker" (Aminoethyoxy-ethoxyethanol-Linker, siehe Abbildung 1) kann am 5'-Ende das Biotin über einen C6-Linker (siehe Abbildung 2), einen TEG-Linker (siehe Abbildung 3) oder einen Prolinol-Linker (siehe Abbildung 4) angekoppelt werden.
Für die Kopplung an das 3'-Ende steht ebenfalls ein Biotin-TEG (Abbildung 5) zur Verfügung, für die Modifikation in der Sequenz ein Biotin -dT (Abbildung 6). Kopplungen am 5'-Ende oder auch intern sind bis zu einer Oligolänge von 140 Basen Länge möglich, 3'-Biotin tragende Oligos bieten wir bis zu einer Länge von 80 Basen an.
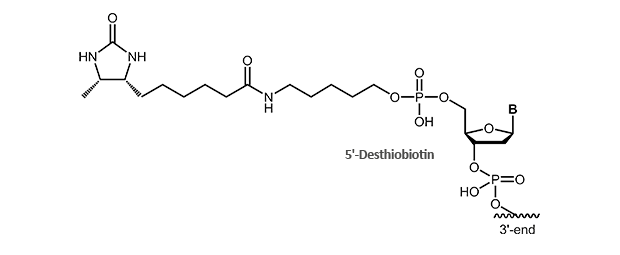
Für eine verstärkte Bindung können zwei Biotine dicht beieinander an das 5'-Ende gekoppelt werden (siehe auch Dual-Biotin). Demgegenüber kann durch die Verwendung von Desthiobiotin-Bausteinen (siehe auch Desthiobiotin) eine abgeschwächte Bindung erreicht werden.
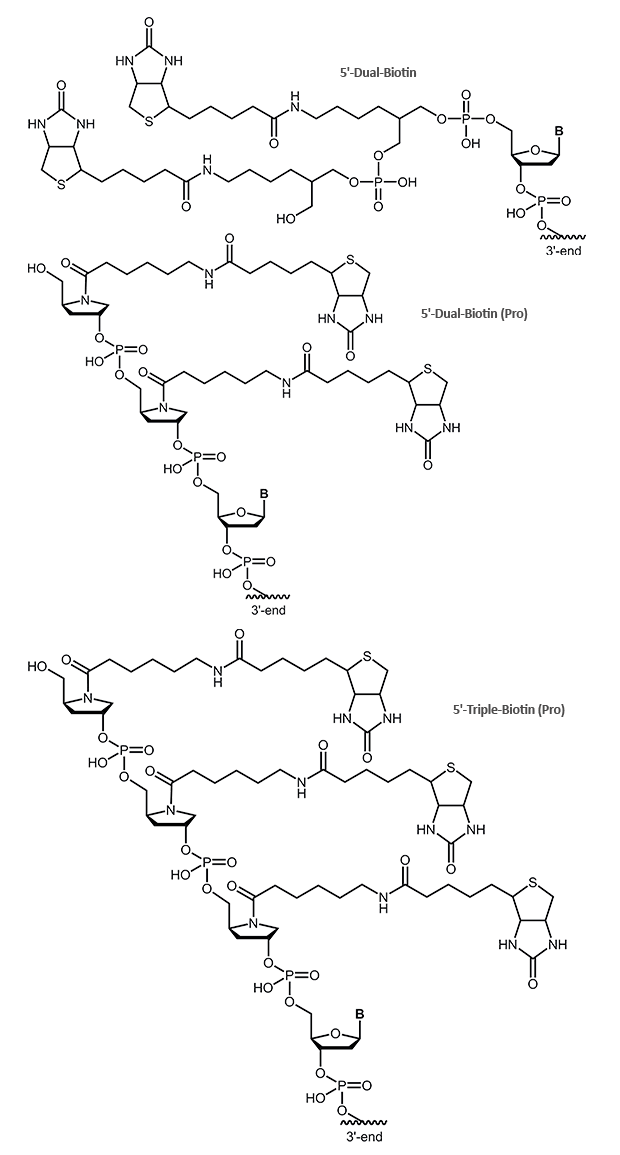
Dual-Biotin, Triple-Biotin
Mit der Dual-Biotin Modifikation werden zwei Biotine an das 5‘-Ende des Oligos gekoppelt. Durch die gleichzeitige, kooperative Bindung beider Biotine an zwei der vier Bindungsstellen des Streptavidins wird die Biotin/Streptavidin-Bindung noch fester. Das Dual-Biotin wird vor allem genutzt, wenn ein thermischer Denaturierungsschritt (z.B. während der PCR) mit Streptavidin-gebundenen Sonden oder Templaten notwendig wird. Dual-Biotin wird in den meisten SAGE Protokollen (Serial Analysis of Gene Expression) verwendet.
Bei der Triple-Biotin Modifikation werden drei Biotine an das 5´-Ende des Oligonucleotids gebunden. Ähnlich dem Dual-Biotion kann diese Biotin-Variante ebenfalls eine festere Bindung zum Streptavidin ausbilden.
Bitte beachten Sie, dass die Online-Bestellung für die Modifikationen 5´-Dual-Biotin (Pro) und 5´-Triple-Biotin (Pro) nicht möglich ist. Bei Interesse wenden Sie sich bitte an unseren Customer Support.
Desthiobiotin
Dem Desthiobiotin fehlt das S-Atom in der Biotin-Ringstruktur. Es bindet dennoch ebenfalls an Streptavidin, aber mit deutlich geringerer Affinität als Biotin. Dies ermöglicht die Reinigung markierter Nukleinsäuren und ihrer Hybride über das Biotin/Streptavidin-System in Austauschreaktion mit Biotin. Da es sich hierbei um ein sehr mildes Verfahren handelt, ist auch die Reinigung von Hybriden oder sensitiven Konjugaten möglich.
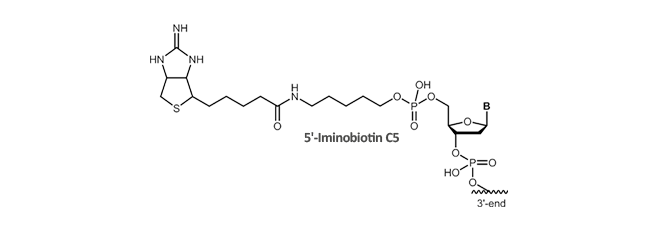
Iminobiotin
Iminobiotin ist ein cyclisches Guanidinanalogon von Biotin und bindet im basischen pH (pH >9) mit hoher Affinität an Avidin oder Streptavidin, zeigt jedoch nur geringe Affinität im sauren pH-Bereich (pH <6). Aufgrund der pH-abhängigen Bindungsaffinität werden Iminobiotin-modifizierte Proteine bei einem pH-Wert von 9 bis 11 auf einer Avidinsäule zurückgehalten und erst bei Änderung des pH-Wertes in den sauren Bereich (pH 4) bzw. durch Zugabe von Biotin eluiert. Im basischen pH-Bereich behält die freie Basenform von Iminobiotin die Bindungseigenschaften von Biotin bei, wohingegen bei niedrigem pH-Wert die Salzform von Iminobiotin nur eine geringere Affinität zu Avidin zeigt, wodurch die Bindung zwischen Avidin und Biotin aufgehoben wird. Dies stellt eine effiziente Methode der Proteinreinigung unter äußerst milden Bedingungen dar und verhindert die Denaturierung gereinigter Proteine.
Literatur:
- Separation of complementary strands of plasmid DNA using the biotin-avidin system and its application to heteroduplex formation and RNA/DNA hybridizations in electron microscopy. Delius H, van Heerikhuizen H, Clarke J, Koller B; Nucleic Acids Res. (1985), 13, 5457-5469.
- In situ hybridization with biotinylated DNA probes: a rapid diagnostic test for adenovirus upper respiratory infections. Gomes SA, Nascimento JP, Siqueira MM, Krawczuk MM, Pereira HG, Russell WC; J. Virol. Methods (1985), 12, 105-110.
- Enhanced RACE method using specific enrichment by biotinylated oligonucleotides bound to streptavidin coated magnetic particles. Lankiewicz S, Gisselmann G, Hatt H; Nucleic Acids Res. (1997), 25, 2037-2038.
- Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations. Dressman D, Yan H, Traverso G, Kinzler KW, Vogelstein B; Proc. Natl. Acad. Sci. USA. (2003), 100, 8817-8822.
- Direct labeling of RNA with multiple biotins allows sensitive expression profiling of acute leukemia class predictor genes. Cole K, Truong V, Barone D, McGall G; Nucleic Acids Res. (2004), 32, e86.
- Serial analysis of ribosomal sequence tags (SARST): a high-throughput method for profiling complex microbial communities.Neufeld JD, Yu Z, Lam W, Mohn WW; Environ. Microbiol. (2003), 6,131-144.
- Molecular interactions on microarrays. Southern EM, Mir K, Shchepinov MS; Nat. Genet. (1999), 21, 5-9.
- Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: uses for protein labeling, detection, and isolation. Hirsch JD, Eslamizar L, Filanoski BJ, Malekzadeh N, Haugland RP, Beechem JM, Haugland RP; Anal Biochem. (2002), 308, 343-57.
- Unbiased proteomic analysis of proteins interacting with the HIV-1 5′LTR sequence: role of the transcription factor Meis. Tacheny A, Michel S, Dieu M, Payen L, Arnould T, Renard P; Nucleic Acids Res. (2012), 40,1-18.
- Robust definition and identification of multiple oomycetes and fungi in environmental samples by using a novel cleavable padlock probe-based ligation detection assay. van Doom R, Slawiak M, Szemes M, Dullemans AM, Bonants P, Kowalchuk GA, Schoen CD; Appl. Environ. Microbiol. (2009), 75, 4185-4193.
- The Use of the 2-Iminobiotin-Avidin Interaction for the Selective Retrieval of Labeled Plasma Membrane Components. Orr GA; The Journal of biological chemistry (1981), Vol. 256, No. 2, Issue of January 25, pp. 761-766.
- Iminobiotin affinity columns and their application to retrieval of streptavidin. Hofmann K, Wood SW, Brinton CC, Montibeller JA, Finn FM; Proc Natl Acad Sci U S A (1980), 77(8): 4666–4668, doi: 10.1073/pnas.77.8.4666.
Digoxigenin (DIG)
Oligonucleotide mit Digoxigenin-Modifikation
Das Steroid Digoxigenin aus der Pflanze Digitalis purpurea, vor etwa 20 Jahren als nicht-radioaktive Markierung für Nukleinsäuren eingeführt, hat sehr breite Akzeptanz erreicht und findet mittlerweile in fast allen Hybridisierungsmethoden (Southern, Northern, ISH or FISH) als Marker für DNA oder RNA Sonden Verwendung.
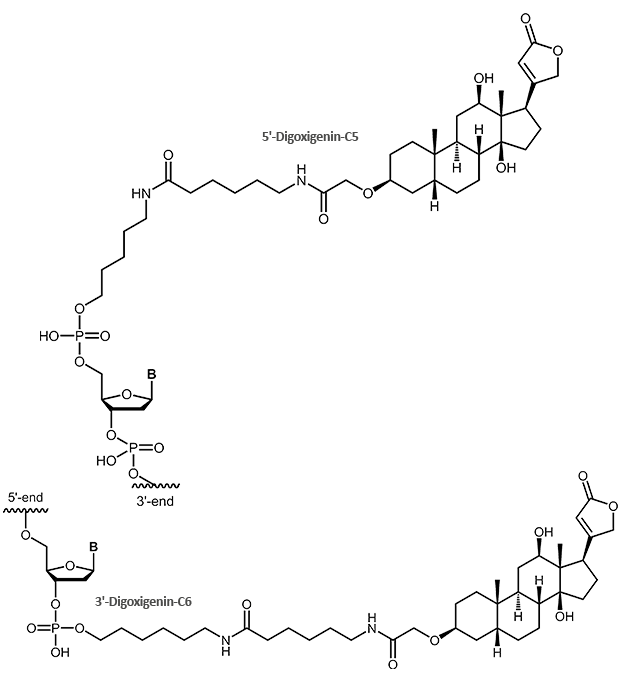 |
Literatur:
- Digoxigenin as an alternative probe labeling for in situ hybridization. Komminoth P; Diagn Mol Pathol. (1992), 1, 142-150.
- Digoxigenin-end labeled oligonucleotides facilitate the detection of minimal residual disease in acute lymphoblastic leukemia. Nakao M, Janssen JWG, Schäfer M, Bartram CR; Leukemia (2000), 14, 767-772.
- Rapid chemiluminescent detection of mRNAs on northern blots with digoxigenin end-labelled oligonucleotides. Trayhurn P, Duncan JS, Nestor A, Thomas ME, Eastmond NC, Rayner DV; Electrophoresis (1995), 16, 341-344.
Dinitrophenol (DNP)
DNP
Das kleine Molekül Dinitrophenol ist seit langem als Marker in der Immunohistochemie und Immunocytochemie etabliert. Es wird auch als Markierung von Oligonukleotid-Sonden genutzt, insbesondere bei Verwendung für die in situ Hybridisierung (ISH oder multicolor FISH).
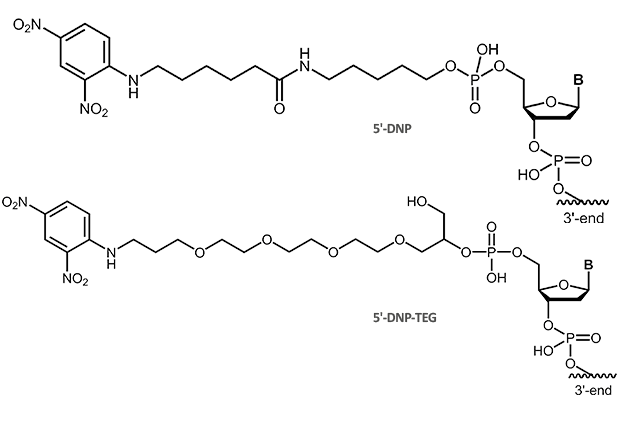 |
Literatur:
- Synthesis and antibody-mediated detection of oligonucleotides containing multiple 2,4-dinitrophenyl reporter groups. Grzybowski J, Will DW, Randall RE, Smith CA, Brown T; Nucleic Acids Res. (1993), 21, 1705-1712.
- A comparative study of digoxigenin, 2,4-dinitrophenyl, and alkaline phosphatase deoxyoligonucleotide labels in non-radioisotopic in situ hybridization. Harper SJ, Bailey E, McKeen CM, Stewart AS, Pringle JH, Feehally J, Brown T, Bright R; J. Clin. Pathol. (1997), 5, 686-690.
Fluorescein (Fam, FITC)
Fluorescein
Der Farbstoff Fluorescein kann nicht nur als Modifikation zur direkten Fluoreszenzmarkierung genutzt werden. Er findet auch als Hapten-Markierung zur Immunodetektion Verwendung, meist in Verbindung mit Farb- oder Chemiluminiszenz-Nachweismethoden. Die Fluorescein-Markierung ist als 6-Fam oder FITC möglich, beide Modifikationen unterscheiden sich nur in der Struktur des Linkers zum Oligonukleotid.
|
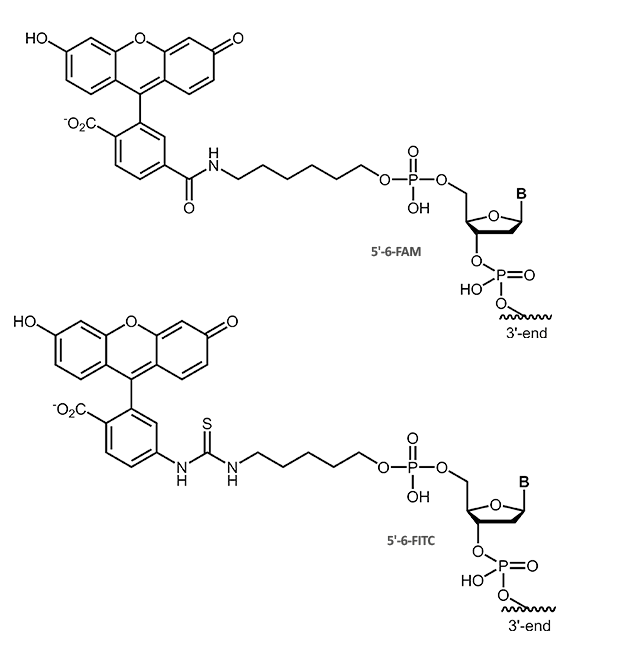 |
Literatur:
- 3’-End fluorochromized and haptenized oligonucleotides as in situ hybridization probes for multiple simultaneous RNA detection. Dirks RW, Van Gijlswijk RPM, Vooijs MA, Smit AB, Bogerd J, Van Minnen J, Raap AK, Van der Ploeg M; Exp. Cell Res. (1991), 194, 310-315.
- Analysis of microRNA expression by in situ hybridization with RNA oligonucleotide probes. Thompson RC, Deo M, Turner DL; Methods (2007), 43, 153-161.
- In situ hybridization with fluoresceinated DNA. Wiegant, J, Ried T, Nederlof PM, Van der Ploeg M, Tanke HJ, Raap AK; Nucleic Acids Res. (1991), 19, 3237–3241.