
DE
|
EN
> Modifizierte Nucleotide (Basen)
N-POM Caged-dT (Internal modification)
N-POM caged dT – Photoaktivierende Modifikation
Um die Funktion und Wirkung eines Biomoleküls zu erforschen, ist es oft hilfreich, wenn die natürliche Funktion gestört werden kann. Dies geschieht idealerweise ohne dass die Störung selbst bereits Nebenwirkungen hervorruft. Eine mögliche und äußerst elegante Herangehensweise ist dabei das sogenannte „Photocaging“.
Hierbei lagert sich an Nucleinsäuren oder auch Peptide eine photolabile Schutzgruppe an, die die natürliche Funktion des Biomoleküls sterisch behindert (caging =einsperren), sodass das Molekül inaktiv wird. Dieses sogenannte „caging“ kann beispielsweise die Hybridisierung komplementärer Basen inhibieren oder die Ausbildung der Sekundärstruktur stören.
Durch Bestrahlung mit Licht einer bestimmten Wellenlänge kann diese Schutzgruppe gezielt abgespalten werden (decaging), wodurch das Originalmolekül wiederhergestellt wird und seine biologische Funktion aufnehmen kann. Die Abspaltung mit Licht erlaubt eine präzise zeitliche und räumliche Regulierung biologischer Prozesse, darüber hinaus ist die Lichtbestrahlung eine minimal-invasive Technik, bei der kaum Nebenwirkungen zu befürchten sind.
Eine bekannte photolabile Schutzgruppe ist das 6-Nitropiperonyloxymethyl (N-POM). Oligonucleotide können an Thyminen mit N-POM modifiziert werden (Abb. 1). Durch die Anknüpfung der N-POM-Gruppe direkt am Pyrimidin-Ring der Base kann das so modifizierte Thymin nicht mehr mit einem komplementären Gegenstrang hybridisieren. Untersuchungen konnten zeigen, dass ein Oligonucleotid mit N-POM-Modifizierungen alle 5-6 Basen in der Sequenz bereits keine Duplexbildung mehr zeigt.
Durch Bestrahlung mit Licht bei einer Wellenlänge von 365 nm kann die N-POM-Gruppe wieder abgespalten und die biologische Aktivität des Oligos vollständig wiederhergestellt werden.
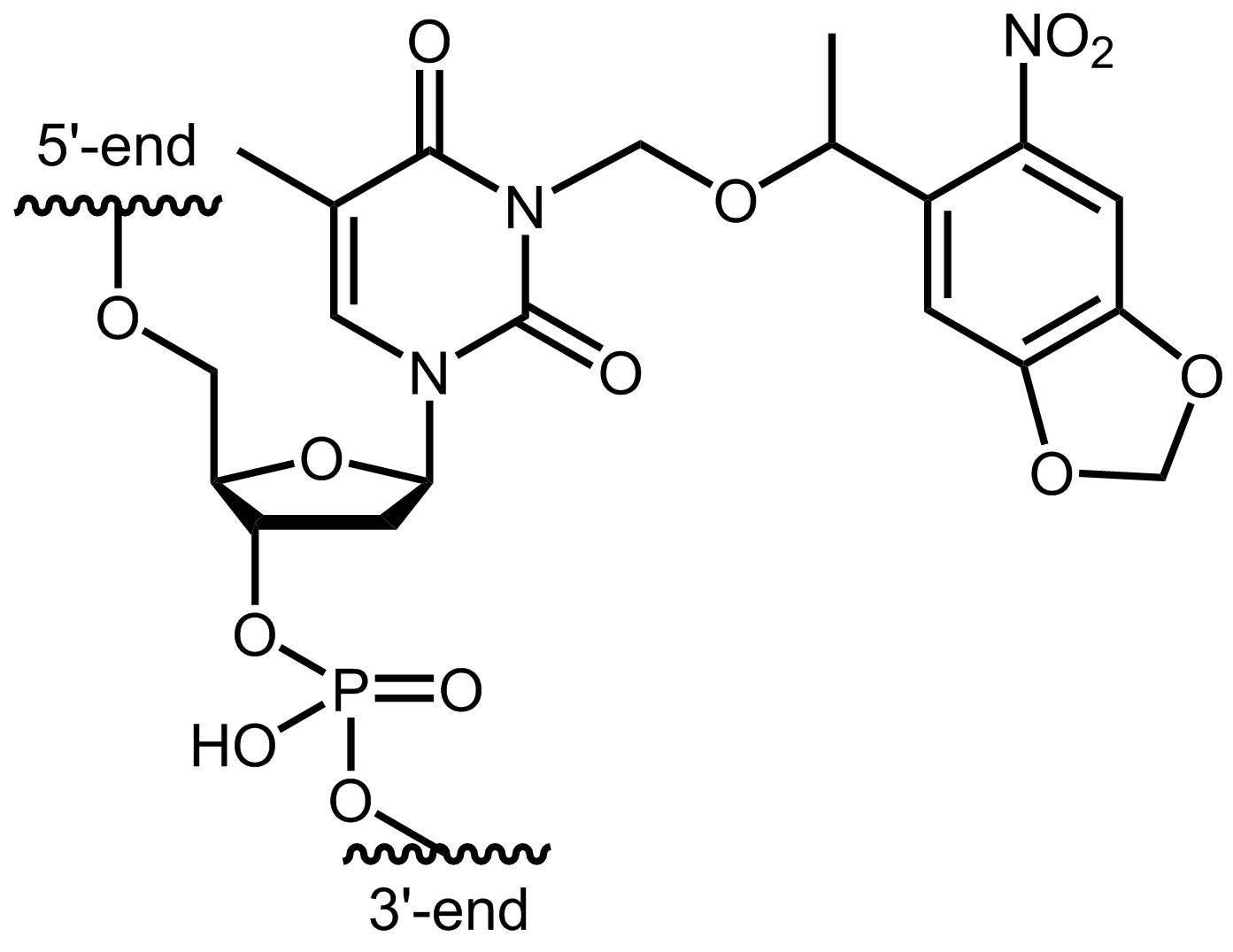
Abbildung 1: Struktur der internen Modifikation N-POM caged dT gebunden an ein Oligo.

Abbildung 2) zeigt die Maldi-Analyse eines N-POM caged dT modifizierten dT21-Oligonucleotids mit einem Molekulargewicht von 6556 Da (grün unterlegt). Der kleinere, linke Peak stellt ein Nebenprodukt dar, das bei dem partiellen Abbau der N-POM Einheit entsteht.
Für eine vollständige Abspaltung der photolabilen Schutzgruppe (Decaging) und damit zur Wiederherstellung der biologischen Aktivität wird das Oligo 30 min bei einer Wellenlänge von 365 nm bestrahlt.

Abbildung 3) zeigt das dT21-Oligonucleotid nach einer 30 minütigen UV-Exposition bei 365 nm. Die N-POM Schutzgruppe fällt daraufhin vollständig vom Oligo ab (MW 6328 Da) (blau unterlegt). Zum Vergleich das N-POM dT-modifizierte dT21-Oligo in grün (MW 6556 Da).
Literatur:
[1] Photoremovable Protecting Groups in Chemistry and Biology: Reaction Mechanisms and Efficacy. Klán P, Šolomek T, Bochet CG, Blanc A, Givens R, Rubina M, Popik V, Kostikov A, Wirz J; Chem. Rev. (2013), 113, 1, 119-191.
[2] Photochemical DNA Activation. Lusic H, Young DD, Lively MO, Deiters A; Org Lett. (2007), 9, 1903–1906.
[3] Restriction enzyme-free mutagenesis via the light regulation of DNA polymerization. Young DD, Lusic H, Lively MO, Deiters A; Nucleic Acids Research (2009), 37, 8, e58.
[4] A New Photocaging Group for Aromatic N-Heterocycles. Lusic H, Deiters A; SYNTHESIS (2006), No. 13, pp 2147–2150.
Um die Funktion und Wirkung eines Biomoleküls zu erforschen, ist es oft hilfreich, wenn die natürliche Funktion gestört werden kann. Dies geschieht idealerweise ohne dass die Störung selbst bereits Nebenwirkungen hervorruft. Eine mögliche und äußerst elegante Herangehensweise ist dabei das sogenannte „Photocaging“.
Hierbei lagert sich an Nucleinsäuren oder auch Peptide eine photolabile Schutzgruppe an, die die natürliche Funktion des Biomoleküls sterisch behindert (caging =einsperren), sodass das Molekül inaktiv wird. Dieses sogenannte „caging“ kann beispielsweise die Hybridisierung komplementärer Basen inhibieren oder die Ausbildung der Sekundärstruktur stören.
Durch Bestrahlung mit Licht einer bestimmten Wellenlänge kann diese Schutzgruppe gezielt abgespalten werden (decaging), wodurch das Originalmolekül wiederhergestellt wird und seine biologische Funktion aufnehmen kann. Die Abspaltung mit Licht erlaubt eine präzise zeitliche und räumliche Regulierung biologischer Prozesse, darüber hinaus ist die Lichtbestrahlung eine minimal-invasive Technik, bei der kaum Nebenwirkungen zu befürchten sind.
Eine bekannte photolabile Schutzgruppe ist das 6-Nitropiperonyloxymethyl (N-POM). Oligonucleotide können an Thyminen mit N-POM modifiziert werden (Abb. 1). Durch die Anknüpfung der N-POM-Gruppe direkt am Pyrimidin-Ring der Base kann das so modifizierte Thymin nicht mehr mit einem komplementären Gegenstrang hybridisieren. Untersuchungen konnten zeigen, dass ein Oligonucleotid mit N-POM-Modifizierungen alle 5-6 Basen in der Sequenz bereits keine Duplexbildung mehr zeigt.
Durch Bestrahlung mit Licht bei einer Wellenlänge von 365 nm kann die N-POM-Gruppe wieder abgespalten und die biologische Aktivität des Oligos vollständig wiederhergestellt werden.

Abbildung 1: Struktur der internen Modifikation N-POM caged dT gebunden an ein Oligo.

Abbildung 2) zeigt die Maldi-Analyse eines N-POM caged dT modifizierten dT21-Oligonucleotids mit einem Molekulargewicht von 6556 Da (grün unterlegt). Der kleinere, linke Peak stellt ein Nebenprodukt dar, das bei dem partiellen Abbau der N-POM Einheit entsteht.
Für eine vollständige Abspaltung der photolabilen Schutzgruppe (Decaging) und damit zur Wiederherstellung der biologischen Aktivität wird das Oligo 30 min bei einer Wellenlänge von 365 nm bestrahlt.

Abbildung 3) zeigt das dT21-Oligonucleotid nach einer 30 minütigen UV-Exposition bei 365 nm. Die N-POM Schutzgruppe fällt daraufhin vollständig vom Oligo ab (MW 6328 Da) (blau unterlegt). Zum Vergleich das N-POM dT-modifizierte dT21-Oligo in grün (MW 6556 Da).
Literatur:
[1] Photoremovable Protecting Groups in Chemistry and Biology: Reaction Mechanisms and Efficacy. Klán P, Šolomek T, Bochet CG, Blanc A, Givens R, Rubina M, Popik V, Kostikov A, Wirz J; Chem. Rev. (2013), 113, 1, 119-191.
[2] Photochemical DNA Activation. Lusic H, Young DD, Lively MO, Deiters A; Org Lett. (2007), 9, 1903–1906.
[3] Restriction enzyme-free mutagenesis via the light regulation of DNA polymerization. Young DD, Lusic H, Lively MO, Deiters A; Nucleic Acids Research (2009), 37, 8, e58.
[4] A New Photocaging Group for Aromatic N-Heterocycles. Lusic H, Deiters A; SYNTHESIS (2006), No. 13, pp 2147–2150.

